细菌抵御病毒侵染Type I R-M系统的最新研究进展
2017年10月2日,PNAS 在线发表了中科院生物物理研究所梁栋材院士课题组的研究论文 “Structural basis underlying complex assembly and conformational transition of the Type I R-M system”。该项工作报道了细菌TypeI系统中核心酶MTase复合物3.2?的晶体三维结构,揭示了MTase在Type I分子机器中识别外源DNA,发挥核心酶活性的分子基础。
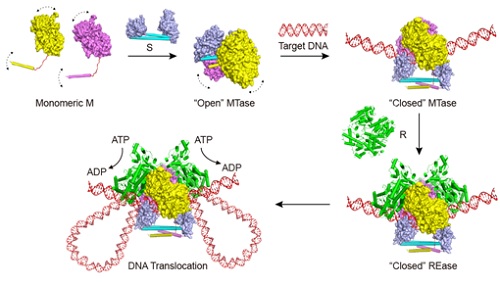
细菌进化一系列的防御系统用来抵御噬菌体病毒的侵染。近年来研究表明,R-M系统和CRISPR-Cas系统同时存在于细菌中。它们作为防御系统的核心,通过直接作用和降解侵入宿主细胞的外源DNA,从而为寄主提供了先天适应性免疫保护。R-M系统存在于90%的细菌中,根据亚基的组成和DNA的切割方式主要划分为Type I,Type II,Type III和Type IV四种。TypeI R-M系统由多个亚基组成,包括DNA识别S-亚基、甲基化M-亚基和限制性内切酶 R-亚基。尽管在过去的五十年中,这个领域有大量的研究报道,但是因为一直没有高分辨率的结构数据,TypeI中各个亚基之间的组装和构象变化并不清楚。
梁栋材课题组闫小雪副研究员在对细菌防御系统长期研究的基础上,与感染免疫中心高璞研究员通力合作,首次报道了TypeI系统中MTase复合物(2M+1S)在小分子SAM存在条件下与双链DNA的三元大复合物“Open”模式的原子分辨率晶体结构。该研究揭示了M与S亚基分子间相互作用借助于一种新型四螺旋束结构域,这个结构域决定了M,S和DNA之间相互作用的特异性;此外,“Open”模型与之前发表的电镜18? “Close”模型有显著的构象变化,生化结果表明R亚基更倾向与“Close”模式的MTase复合物结合。基于这些结果,我们提出了Type I R-M分子机器组装的新模型。该研究对于深入理解细菌抵御病毒入侵的Type I R-M系统的分子机制具有重要的意义,这一工作为TypeI限制修饰系统今后在分子生物学中的应用提供了有力的结构和理论依据。
闫小雪副研究员和高璞研究员为本文的共同通讯作者;梁栋材课题组助理研究员刘晏平为本文的第一作者;唐群副研究员、张介中硕士,田利飞博士为主要完成人。该项研究得到了国家自然科学基金、中国科学院战略性先导科技专项的资助,上海同步辐射光源和蛋白质科学研究平台为该研究提供了重要的技术支持。
图示:TypeI R-M系统分子机器模型
(供稿:梁栋材课题组)
附件下载: