
单碱基编辑器的脱靶效应及预测工具
在人类致病性遗传变异中,单核苷酸变异(single nucleotide variant, SNV)占比约为58%,SNV中61%是"转换"类型[1]。随着人们对基因组DNA序列影响人类健康的认识不断增加,基因组编辑技术应用于临床治疗的潜力已经显现出来。这也让人类的终极梦想--"随心所欲"地精确编辑基因--变得不再遥不可及。
CRISPR-Cas(clustered regularly interspaced short palindromic repeats and CRISPR associated)是一种天然存在于细菌和古细菌中用于抵御病毒入侵的防御机制,因其向导RNA的可编程特性和Cas核酸酶在多种细胞和组织中的明显活性而被广泛地用于基因组编辑。CRISPR-Cas系统进行基因组编辑的方式是在细胞内目标编辑的位点引入双链DNA断裂(DSB)。然而,CRISPR-Cas系统引入的DSB会对基因组稳定性产生不良影响,需要更可靠的基因组编辑技术进行精确的DNA或RNA编辑。
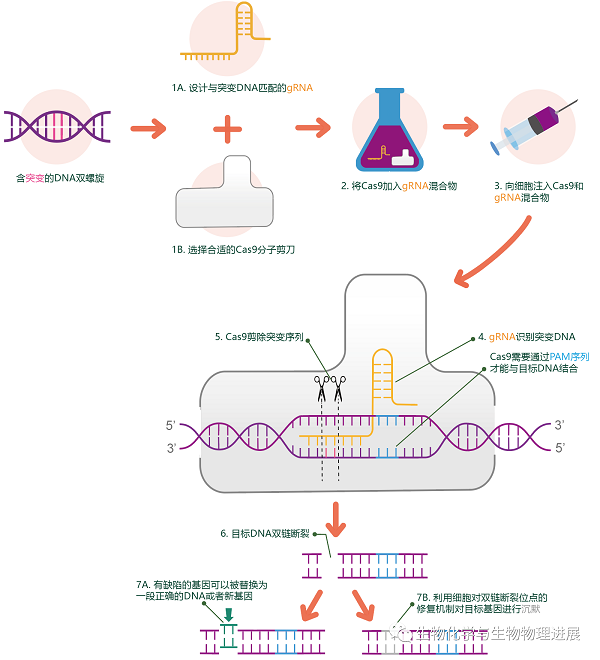
CRISPR-Cas基因编辑技术说明
单碱基编辑器
基于CRISPR-Cas系统的单碱基编辑器自2016年诞生以来就显示出广阔的临床应用前景。单碱基编辑器(base editors, BEs)能高效地对指定位点的SNV进行精准修复;不同于Cas9在编辑位点产生DSB,BEs不会引入DSB,从而被认为是更安全的基因组编辑工具,为治疗遗传性疾病提供了良好方案[2]。主流的BEs由催化活性受损的Cas9酶(nCas9)连接脱氨酶构成,在单向导RNA(single-guide RNA, sgRNA)的引导下,定位到预先设计的基因组位置发挥作用。nCas9能识别一种被称为原间隔序列邻近基序(protospacer adjacent motif, PAM)的序列,sgRNA所靶向的DNA片段通常在PAM上游。sgRNA与DNA序列的互补配对使得DNA双链打开,形成R-loop结构。此时暴露出的非靶向DNA单链可以作为脱氨酶的底物,在脱氨酶编辑范围内(编辑窗口)相应类型的碱基被脱氨基,进一步通过DNA修复过程实现类型转换。根据脱氨酶实现的单碱基替换类型,BEs可分为腺嘌呤碱基编辑器(ABEs)和胞嘧啶碱基编辑器(CBEs)。ABEs能实现A到G的直接转换,CBEs能将C转换为T。综合ABEs和CBEs,它们能实现对所有转换类型的SNV的修复。目前临床实践中,已经实现了在细胞系或小鼠中对致病性单核苷酸突变的纠正。例如,改善β血红蛋白病、β地中海贫血和镰刀型细胞贫血症等单基因疾病[3-4];恢复患有Leber氏先天性黑蒙症(一种遗传性视网膜疾病)成年小鼠的视觉功能[5]。在肿瘤免疫治疗中,将单碱基编辑系统用于对嵌合抗原受体(CAR)-T细胞的离体改造同样具有光明的治疗前景[6]。
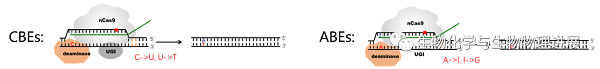
CBEs和ABEs两种BEs的基因编辑模式图
不容忽视的非靶向编辑
作为一种治疗手段,基因组编辑工具必须具有较高的靶向编辑效率和最少的有害或非靶向编辑。
根据已有的研究,BEs可能在基因组上与靶向位点序列相似的其他位点发生脱靶编辑,这种由序列相似引起的脱靶被称为"sgRNA依赖型脱靶"。基于大量的CRISPR-Cas9系统高通量实验, 已经开发出多种基于不同原理的预测工具。单碱基编辑器可以结合到基因组上与目标原间隔序列具有高度序列同源性的脱靶位点上。BEs在这些sgRNA依赖的脱靶位点上可以发生单碱基编辑。
除了sgRNA依赖的脱靶编辑,还有一种类型的脱靶是"sgRNA非依赖型脱靶",由BEs上的脱氨酶结构域与DNA结合引入。中科院神经科学研究所杨辉实验室和中科院遗传与发育生物学研究所高彩霞实验室的研究人员在2019年报道了CBEs中的BE3在小鼠胚胎和水稻中过表达会带来随机的基因组范围内的突变,其平均数量分别为5×10-8/bp和5.3×10-7/bp [7-8]。这些脱靶编辑可能由BE3的脱氨酶结构域天然的对DNA的亲和性导致,并不依赖于sgRNA指导下Cas9与DNA的结合。有研究随后证明了CBEs可以在人诱导多能干细胞中引入sgRNA非依赖的脱靶突变[9]。与sgRNA依赖的脱靶编辑不同,sgRNA非依赖的脱氨作用发生在细胞基因组上的不同位置,导致不能通过靶向高通量测序鉴定脱靶位点。
不同的研究比较了CBEs和ABEs的sgRNA非依赖型脱靶活性,均发现CBEs的脱靶频率高于ABEs[10]。修饰脱氨酶结构域或者使用其他类型的脱氨酶可以得到具有较低sgRNA非依赖型DNA脱靶水平并且靶向编辑效率依然较高的CBEs变体(如YE1-BE4、R33A-BE4、YE1-BE4-CP1028、YE1-BE4-NG、AmAPOBEC1、SsAPOBEC3B、YE1-BE3、 FE1-BE3)[11]。
一种可选用的实现高靶向编辑效率、低脱靶效率的方法是将BEs以mRNA或者核糖核蛋白的形式导入细胞,该方法缩短了BEs在细胞中的暴露时长,可以减少sgRNA非依赖的脱靶编辑。类似地,将BEs以mRNA的形式递送到细胞可以通过减少BEs表达时长来减少sgRNA非依赖的脱靶编辑。因此,相较于质粒转染和病毒转导,瞬转BEs核糖核蛋白或者mRNA有利于减少脱靶效应。
脱靶效应的评估方法和预测工具
现有的评估CRISPR-Cas系统在真核细胞中的特异性的方法主要有三类:基于计算的工具(如Cas-OFFinder[12]、CFD[13]、BEdeepoff[14])、体内或体外捕获DNA双链断裂位点的方法(如GUIDE-seq、DISCOVER-seq)、体外片段化基因组DNA的方法(如Digenome-seq[15]、CIRCLE-seq)。
基于计算的方法是根据靶向位点和脱靶位点的序列相似性来预测潜在的脱靶位点。后两类方法都无法评估BEs在体内的特异性,因为不同于CRISPR-Cas系统在编辑基因组时需要引入DNA双链断裂,BEs很少会造成DNA双链断裂,BEs的主要产物是SNV,而SNV是以上两类方法所无法捕获的。为了弥补以上两类方法对于检测BEs脱靶效应的不足,研究人员开发出基于单细胞、单克隆技术的全基因组测序(whole genome sequencing, WGS)方法,该方法为体内检测全基因组范围内的SNV和INDEL提供了可能,WGS通过比较编辑后的基因组与对照组存在的变异差异,确定由基因组编辑工具造成的变异。但WGS成本高昂且难以实现对BEs脱靶效应的高通量检测。最新开发出了可以灵敏、高效地评估CBEs在全基因组水平脱靶效应的方法--Detect-seq[2]。研究人员利用CBEs在进行编辑过程中产生的中间产物脱氧尿嘧啶(dU),对CBEs产生的dU进行生物素标记后富集;同时在dU位点附近,将正常的胞嘧啶C替换为d5fC,随后通过化学标记反应,产生串联的C转换为T的信号,从而增加了CBEs脱靶检测的灵敏度。利用该技术,发现了两种新的sgRNA依赖型的脱靶:在原间隔序列外的编辑(out-of-protospacer edit)和靶向链编辑(target-strand edit)。
sgRNA非依赖型脱靶可以通过优化BEs的脱氨酶,提高BEs特异性将这类脱靶降低到背景水平。sgRNA依赖型脱靶是由基因组内存在目标位点以外的,与sgRNA序列相似的其他位点导致的。由于真核生物基因组中存在上百个其他位点与目标编辑序列相似,如何降低sgRNA依赖型脱靶仍是一个充满挑战的课题[14]。据我们所知,目前已有的预测BEs的sgRNA依赖型脱靶的工具仅有BEdeepoff,在BEdeepoff开发出来以前,预测BEs脱靶的普遍方法是参考CRISPR/Cas系统的脱靶预测工具。
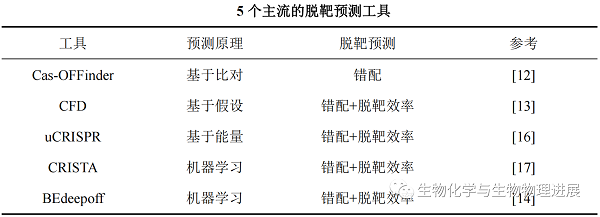
由于BEs间活性窗口、编辑活性、靶向序列偏好性等性质的不同,目前已有的脱靶预测工具拓展应用于其他类型的BEs的准确性有待进一步验证。为了弥补当前对全面评估BEs脱靶效应的工具的缺乏,北京协和医学院基础学院王晓月课题组开发了单碱基编辑设计和脱靶评估工具BE-dot(详情请点击阅读原文)。BE-dot能对目标编辑的SNV设计编辑方案,并从编辑效率、脱靶图谱、脱靶带来的功能影响等方面对编辑方案综合评估,期望以此为生物医学研究提供便利。在脱靶图谱预测中,BE-dot综合了多个预测工具,如Cas-OFFinder、CALITAS、CFD、uCRISPR以及最新开发的BEdeepoff,能对Cas9依赖型脱靶提供较全面的评估,有利于确定脱靶热点。
展望
未来可以凭借更多单碱基编辑实验探索sgRNA-DNA不同错配情况影响脱靶编辑效率的一般规律。目前针对BEs脱靶效应的预测工具,相应工具的开发仍然有较大的空白。新的高通量评估BEs脱靶的实验方法的开发,如Detect-seq、sgRNA-脱靶位点报告系统,以及高精度检测低频突变的实验方法都为高效、经济地评估脱靶带来新的可能。
参考文献
[1] Rabinowitz R, Abadi S, Almog S, et al. Prediction of synonymous corrections by the BE-FF computational tool expands the targeting scope of base editing. Nucleic Acids Res, 2020, 48(W1): W340-W347
[2] Lei Z, Meng H, Lv Z, et al. Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors. Nat Methods, 2021, 18(6): 643-651
[3] Liang P, Ding C, Sun H, et al. Correction of β-thalassemia mutant by base editor in human embryos. Protein Cell, 2017, 8(11): 811-822
[4] Antoniou P, Miccio A, Brusson M. Base and prime editing technologies for blood disorders. Front Genome Ed, 2021, 3: 618406
[5] Suh S, Choi E H, Leinonen H, et al. Restoration of visual function in adult mice with an inherited retinal disease via adenine base editing. Nat Biomed Eng, 2021, 5(2): 169-178
[6] Liu X, Zhang Y, Cheng C, et al. CRISPR-Cas9-mediated multiplex gene editing in CAR-T cells. Cell Res, 2017, 27(1): 154-157
[7] Zuo E, Sun Y, Wei W, et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science, 2019, 364(6437): 289-292
[8] Jin S, Zong Y, Gao Q, et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science, 2019, 364(6437): 292-295
[9] Mcgrath E, Shin H, Zhang L, et al. Targeting specificity of APOBEC-based cytosine base editor in human iPSCs determined by whole genome sequencing. Nat Commun, 2019, 10(1): 5353
[10] Lee H K, Smith H E, Liu C, et al. Cytosine base editor 4 but not adenine base editor generates off-target mutations in mouse embryos. Commun Biol, 2020, 3(1): 19
[11] Doman J L, Raguram A, Newby G A, et al. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nat Biotechnol, 2020, 38(5): 620-628
[12] Bae S, Park J, Kim J-S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014, 30(10): 1473-1475
[13] Doench J G, Fusi N, Sullender M, et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat Biotechnol, 2016, 34(2): 184-191
[14] Zhang C, Wang D, Qi T, et al. BEdeepoff: an in silico tool for off-target prediction of ABE and CBE base editors. bioRxiv, 2021, 2021.2003.2014.435296
[15] Kim D, Kim D-E, Lee G, et al. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nat Biotechnol, 2019, 37(4): 430-435
[16] Zhang D, Hurst T, Duan D, et al. Unified energetics analysis unravels SpCas9 cleavage activity for optimal gRNA design. Proc Natl Acad Sci USA, 2019, 116(18): 8693-8698
[17] Abadi S, Yan W X, Amar D, et al. A machine learning approach for predicting CRISPR-Cas9 cleavage efficiencies and patterns underlying its mechanism of action. PLoS Comput Biol, 2017, 13(10): e1005807
作者简介
王泽鲁:北京协和医学院基础学院硕士,研究方向为生物信息学与功能基因组学。
(作者:王泽鲁)
(本文来源于公众号:生物化学与生物物理进展)
附件下载: