
力挽狂澜的RNA聚合酶--DNA损伤修复的最后关卡
生物体在正常生命过程中会面临内/外因来源的DNA损伤,DNA损伤不仅影响基因的正确复制,也阻碍其正常转录。为避免DNA损伤带来的灾难性后果,生物体进化出一整套修复机制,以保证复制和转录的正确性、基因组的完整性和遗传的稳定性。常见的修复方式有光激活修复系统、错配修复、剪切修复、同源重组修复等。值得注意的是,基因转录过程也是独特的DNA损伤修复机制。
多亚基蛋白复合体RNA聚合酶(RNA polymerase,RNAP)是完成基本生命活动的一员"大将",保守存在于细菌、古细菌、真核生物中,负责转录合成各类RNA,其核心酶发挥主要的合成作用。细菌RNAP核心酶结构最为简单,古细菌核心酶与真核生物RNAPⅡ有显著的结构保守性。真核生物中的RNAPⅠ、RNAPⅡ、RNAPⅢ核心酶结构具有同源性,但分别发挥不同的功能,其中RNAPⅡ负责转录所有编码蛋白的基因和许多非编码RNA。

转录过程中,RNAP沿模板链的行进路途并非一帆风顺,基因组DNA总是不可避免的遭受内外环境带来的损伤。转录模板链上的DNA损伤,如单链断裂、双链断裂等会阻碍RNAP在模板链上的正常前行【1】。
研究发现,当转录中的DNA双链产生诱变损伤时,转录模板链的修复程度要高于编码链,模板链的修复比编码链修复更快,而编码链的修复与基因组DNA的修复节奏基本一致,这说明转录中优先修复模板链中的DNA损伤【2】。RNAP沿模板DNA转录过程中,会感知DNA损伤,并招募修复蛋白,继而修复损伤DNA【3】,此过程称为RNAP监视(RNA polymerase-surveilled,RNAP-S)的DNA修复。

RNAP在参与转录过程中受到阻碍【4】
RNAPⅡ在感知DNA损伤时,不与受损的碱基直接发生作用,而是感知其转录发生障碍后引起的空间位阻【4】。RNAPⅡ结构中的桥螺旋(bridge helix,BH)负责连接RNAPⅡ的两个部分,将RNAPⅡ催化位点与下游的主、次要通道分开,模板装载中越过BH的步骤可作为RNAP变位的检验点。RNAPⅡ装载DNA模板时,DNA下游模板需要越过桥螺旋才能到达活性位点添加NTPs以进行正常转录,此跨越步骤需要模板链发生显著的构象变化。但存在大规模损伤的DNA链往往由于受损的碱基与RNAPⅡ桥螺旋结构上方相结合,而不能发生正常的构象变化,RNAPⅡ无法正常装载DNA模板链,变位步骤受到阻碍,因此发生滞留现象【5】。
RNAP监视下的修复策略
单枪匹马--
当遇到较小的损伤如CPD、AP、Gh时,RNAPⅡ虽然受到阻碍,但不足以被滞留。在这种状态下,RNAPⅡ缓慢的经过损伤位点,并发生不依赖于模板的AMP优先的碱基错误掺入,导致转录产物mRNA中相对损伤的位点的突变,这种易错的修复方式被称为A规则【5】。

团队协作--
RNAP因DNA损伤滞留在DNA模板链上时,会被转录偶联修复因子识别,此时RNAP从模板解离、回溯变位、降解,并引发后续修复蛋白的组装和修复。
例如,细菌中转录偶联因子Mfd,它可以解离模板链上较强损伤处的RNAP同时引发核苷酸切除修过程。有趣的是,它也能促进较弱损伤处RNAP的变位而跨越损伤,Mfd与非NER因子共同作用,以易错的方式修复DNA损伤【6】。原核生物中的DskA也以类似Mfd的方式使DNA损伤处的RNAP发生解离,DNA模板上的损伤进而被修复【7】。Mfd的真核生物同源蛋白CSB同样实施RNAP-S修复,其过程受到更多因素的调控【8】。
原核生物NER系统修复因子之一的Uvrd能直接使滞留的RNAP发生回溯,暴露的损伤部分进而能被修复蛋白修复【9】。真核生物中的OGG1也可引发RNAP-S-BER修复【10】。此外,也有研究显示,RNAPⅢ在同源重组介导的DNA双链断裂修复中发挥了关键的作用【11】。
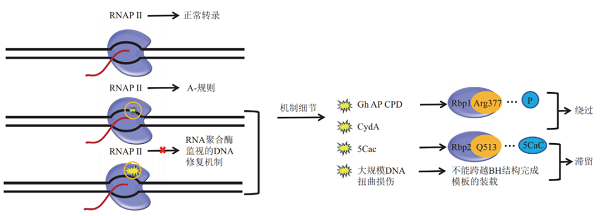
RNAPⅡ监视下的修复策略
RNAP-S偶联DNA修复的生物学意义
确保基因组的稳定--
RNA-S修复对基因组稳定性的维持有重要作用。RNAP在模板DNA损伤处的长期停滞会导致错误碱基掺入,从而导致RNAP-S修复的失败,而基因组不稳定将威胁细胞的存活。但在营养胁迫下发生的易错方式的修复引入的突变却提高了遗传多样性,有利于细胞逃离限制生长的条件,增强了细胞对环境的适应能力【12】。
防御疾病--
RNAP-S修复影响生物体对癌症的预防。例如与RNAP-S修复缺陷相关的柯凯因氏综合征(Cockayne Syndrome,CS)、紫外线敏感综合征(UV-sensitive Syndrome,UVSS)。这两种疾病都是由于相关基因的突变导致RNAP-S-NER缺陷而引起【13-14】。此外,视网膜退行性疾病、范可尼贫血症、肺癌、亨廷顿氏病症等疾病也与RNAP-S修复途径受损有关。
展望
目前人们已经较深入的了解了DNA损伤修复,并揭示了多种DNA损伤修复途径。然而在RNAP监视的DNA修复中,很多机制仍有待于研究。尤其是真核细胞RNAP-S途径的很多细节尚不清楚。但相信随着分子生物学技术手段的革新,这些问题可以被回答。或许在不久的将来我们也可以靶向抑制或加强RNAP-S修复系统来治疗人类不同疾病。(详情请点击阅读原文)
参考文献
[1] Lans H, Hoeijmakers J H J, Vermeulen W, et al. The DNA damage response to transcription stress. Nat Rev Mol Cell Biol, 2019, 20(12): 766-784
[2] Oztas O, Selby C P, Sancar A, et al. Genome-wide excision repair in Arabidopsis is coupled to transcription and reflects circadian gene expression patterns. Nat Commun, 2018, 9(1): 1503
[3] Gregersen L H, Svejstrup J Q. The cellular response to transcription-blocking DNA damage. Trends Biochem Sci, 2018, 43(5): 327-341
[4] Oh J, Xu J, Chong J, et al. Molecular basis of transcriptional pausing, stalling, and transcription-coupled repair initiation. Biochim Biophys Acta Gene Regul Mech, 2021, 1864(1): 194659
[5] Shin J H, Xu L, Wang D. Mechanism of transcription-coupled DNA modification recognition. Cell Biosci, 2017, 7:9
[6] Le T T, Yang Y, Tan C, et al. Mfd dynamically regulates transcription via a release and catch-up mechanism. Cell, 2018, 173(7): 1823
[7] Myka K K, Küsters K, Washburn R, et al. DksA-RNA polymerase interactions support new origin formation and DNA repair in Escherichia coli. Mol Microbiol, 2019, 111(5): 1382-1397
[8] Van Der Weegen Y, Golan-Berman H, Mevissen T E T, et al. The cooperative action of CSB, CSA, and UVSSA target TFIIH to DNA damage-stalled RNA polymerase II. Nat Commun, 2020, 11(1): 2104
[9] Kawale A A, Burmann B M. UvrD helicase-RNA polymerase interactions are governed by UvrD's carboxy-terminal Tudor domain. Commun Biol, 2020, 3(1): 607
[10] Hao W, Wang J, Zhang Y, et al. Enzymatically inactive OGG1 binds to DNA and steers base excision repair toward gene transcription. FASEB J, 2020, 34(6): 7427-7441
[11] Liu S, Hua Y, Wang J, et al. RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell, 2021, 184(5): 1314-1329.e1310
[12] Martin H A, Porter K E, Vallin C, et al. Mfd protects against oxidative stress in Bacillus subtilis independently of its canonical function in DNA repair. BMC Microbiol, 2019, 19(1): 26
[13] Laugel V, Dalloz C, Durand M, et al. Mutation update for the CSB/ERCC6 and CSA/ERCC8 genes involved in Cockayne syndrome. Hum Mutat, 2010, 31(2): 113-126
[14] Ijaz A, Wolf S, Mandukhail S R, et al. UV-sensitive syndrome: whole exome sequencing identified a nonsense mutation in the gene UVSSA in two consanguineous pedigrees from Pakistan. J Dermatol Sci, 2019, 95(3): 113-118
作者简介
王菲尔:中国科学技术大学生物化学与分子生物学专业硕士研究生,主要研究方向为酵母代谢工程。
杨绎煊:南京大学生物化学与分子生物学专业硕士研究生,主要研究方向为病毒感染以及宿主免疫应答过程中的生物大分子结构和功能。
(作者:王菲尔、杨绎煊)
(本文来源于公众号: 生物化学与生物物理进展)
附件下载: