
生物物理所在III-E型 CRISPR-Cas系统的研究方面取得新进展
CRISPR-Cas系统广泛存在于细菌、古菌和某些细菌的病毒中(bacteriophage),它可以特异性识别并降解外源入侵的基因。根据CRISPR-Cas系统干扰机制的不同,这些系统主要被分为两大类(Class1和Class2),其中Class1由多个蛋白构成效应复合物(包括I、III和IV型);Class2由单个蛋白行使功能(包括II、V和VI型)。III型CRISPR-Cas系统进一步可主要分为III-A和III-B两个亚型,它们的特点是具有Cas10蛋白,Cas10通过和Cas5、多个拷贝的Cas11、Cas7以及一条crRNA(CRISPR RNA)组装成多亚基效应复合物。III-A和III-B型CRISPR-Cas系统的多亚基构成在某种程度上限制了这些系统作为基因编辑和检测工具的可操作性。
2020年,Makarova等人报道了一种新型的III型CRISPR-Cas系统,即III-E型CRISPR-Cas系统,生物信息学分析揭示该系统的关键蛋白(称之为Cas7-11或gRAMP)是一个包含有数个Cas7结构域和一个Cas11结构域的单链蛋白【1】。随后的生化实验进一步揭示, Cas7-11能够加工pre-crRNA使之成为成熟的crRNA,并且Cas7-11能够特异性切割单链目标RNA(target RNA,tgRNA)底物。这一生化特点和VI型CRISPR-Cas系统中的Cas13a比较相似(Class2),但是Cas7-11并不显示出Cas13a所具有的底物旁切活性,Cas7-11敲低细胞RNA时也并无明显的细胞毒性,这就使得Cas7-11在被改造成为一款可靠的RNA操控工具方面显示出巨大的潜能。有趣的是,Cas7-11基因附近含有多个CRISPR-Cas相关蛋白,包括Csx29(TPR-CHAT)、Csx30、Csx31和RpoE等。尤其是Csx29,它可与Cas7-11形成稳定的复合物,并能够抑制Cas7-11切割tgRNA的活性。Csx29由N端的TPR结构域和C端的CHAT结构域组成,CHAT结构被认为具有可调控的蛋白酶活性且与细胞程序性死亡相关【2-3】。以上信息揭示III-E型CRISPR-Cas可能具有全新的免疫应答机制,然而,Cas7-11如何加工pre-crRNA、如何识别并切割tgRNA、Csx29如何调节Cas7-11的活性以及Csx29如何被激活等机制并不清楚。为了回答这些科学问题,生物物理所的研究人员针对来自Desulfonema ishimotonii菌的DiCas7-11-crRNA复合物开展了结构与功能研究,并解析了一系列该复合物相关的冷冻电镜结构(图1)。
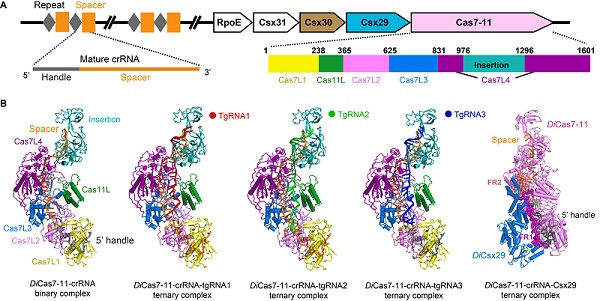
图1. A, Desulfonema ishimotonii III-E型CRISPR-Cas系统的基因组成形式。
B,解析的一系列DiCas7-11-crRNA相关复合物的结构。
研究人员首先解析了DiCas7-11-crRNA未结合底物tgRNA的3.53 Å的冷冻电镜结构。结构分析揭示DiCas7-11-crRNA结构整体呈现'海马'状,DiCas7-11由4个Cas7L(Cas7-like)结构域(Cas7L1-4)、1个Cas11L结构域和1个Insertion结构域组成,一条长38nt的crRNA(15 nt 5' handle + 23 nt spacer)贯穿于整个DiCas7-11蛋白内部。15 nt 5' handle 和Cas7Ll、Cas7L2紧密结合,随后生化实验证实Cas7L1保守的残基His43可切开pre-crRNA的-15和-16碱基之间的磷酸二酯键以生成成熟的crRNA。为了了解DiCas7-11-crRNA如何识别并切割tgRNA,研究团队随后解析了DiCas7-11-crRNA与tgRNA复合物的3.64 Å的冷冻电镜结构。结构分析揭示crRNA和tgRNA形成碱基互补配对,揭示了Cas7L2和Cas7L3切割tgRNA的结构细节。有趣的是,删除Insertion结构域能够增强DiCas7-11切割tgRNA活性的同时并不损害其切割底物的特异性和加工pre-crRNA的能力,这就揭示可以通过删除Insertion结构域可将DiCas7-11改造成为更小的RNA操控工具。研究者进一步解析了DiCas7-11-crRNA和含有与5' handle 配对的 3' anti-tag的tgRNA(tgRNA中与crRNA 5' handle对应的部分称之为"3' anti-tag")复合物的冷冻电镜结构(3.46 Å),以及DiCas7-11-crRNA和含有与5' handle 不配对的 3' anti-tag的tgRNA复合物的冷冻电镜结构(3.14 Å),从结构和生化上揭示tgRNA的3' anti-tag并不参与DiCas7-11-crRNA切割底物活性的调节,这是不同于Cas13a的重要性特性。
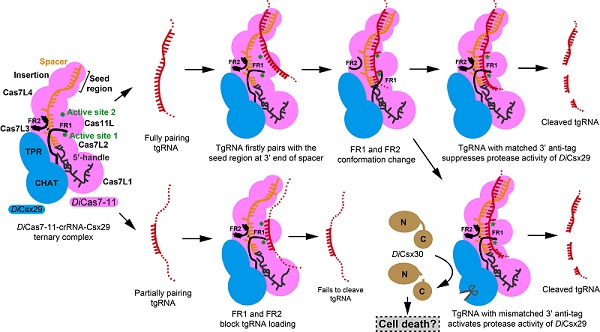
图2. DiCas7-11-crRNA-Csx29活性的调节。
为了研究DiCsx29调节DiCas7-11切割tgRNA的分子机制,研究人员解析了DiCas7-11-crRNA-DiCsx29复合物3.29 Å的电镜结构,发现DiCas7-11-crRNA结构中两段观察不到的区域,即FR1(366-397 aa)和FR2(1316-1337 aa),在结合DiCsx29后变得稳定有序下来。FR1与FR2通过形成一对氢键共同阻碍了tgRNA与crRNA在A1至G9的碱基配对,从而调控了DiCas7-11切割tgRNA活性,这一结论随之得到了生化实验的证实。 此外,DiCas7-11-crRNA-Csx29切割tgRNA同样不依赖于5' handle碱基的配对与否。通过DiCas7-11-crRNA-Csx29与III-A型CRISPR-Cas系统以及相关结构的比较【4-6】,研究人员推测DiCsx29活性的激活可能依赖于tgRNA 3'anti-tag的调控,这一假设得到了该研究后续生化实验的支持。当tgRNA的3' anti-tag与crRNA 5' handle碱基不配对时,DiCsx29蛋白酶活性被激活并切割DiCsx30,被切割后的DiCsx30可能会引起宿主的死亡(图2)。该研究进一步发现,当tgRNA与crRNA 5' handle碱基不配对大于4 nt,并且与spacer碱基配对不少于12 nt时,DiCsx29均可以有效切割DiCsx30。值得一提的是,由于该蛋白的重要性,近期有多个类似的工作发表【7-15】, 与这些工作相比,研究人员在crRNA的3' 端区域充当"种子区域"(seed region)的作用来调节DiCas7-11活性,以及利用FR1与FR2所形成的"离子锁"的别构效应来调节DiCas7-11活性等方面提出了较为新颖的观点(图2)。
综上,该研究详细阐述了DiCas7-11加工pre-crRNA和切割tgRNA的机制、DiCsx29对DiCas7-11切割tgRNA活性调控的机制以及DiCsx29蛋白酶活性激活的调控机制。该研究丰富了人们对CRISPR-Cas系统的理解,有助于将DiCas7-11改造成为一种新型的、无细胞毒性的RNA操控工具。
该项工作于2023年1月26日以题为《Cryo-EM structure and protease activity of the type III-E CRISPR-Cas effector》的论文发表在《Nature Microbiology》。中国科学院生物物理研究所江涛研究员和霍艳高博士为本文共同通讯作者,霍艳高博士和博士研究生赵宏深为共同第一作者,实验师董清华也参与了部分研究工作。该研究得到科技部、中国科学院战略性先导科技专项经费支持。该研究样品的鉴定、冷冻电镜样品的筛查和数据收集工作得到了生物物理研究所蛋白质科学研究平台和生物成像中心等有关工作人员的支持和帮助。
文章连接:https://www.nature.com/articles/s41564-022-01316-4
参考文献:
1. Makarova, K.S. et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbio, l18, 67-83 (2020).
2. Ozcan, A. et al. Programmable RNA targeting with the single-protein CRISPR effector Cas7-11. Nature, 597, 720-725 (2021).
3. Van Beljouw, S.P.B. et al. The gRAMP CRISPR-Cas effector is an RNA endonuclease complexed with a caspase-like peptidase. Science, 373, 1349-1353 (2021).
4. Huo, Y. et al. Cryo-EM structure of Type III-A CRISPR effector complex. Cell Res, 28, 1195-1197, (2018)
5. Kato, K. et al. Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex. Cell 185, 2324-2337 e16 (2022)
6. Hu, C. et al. Craspase is a CRISPR RNA-guided, RNA-activated protease. Science 377, 1278-1285 (2022).
7. Wang, S., Guo, M., Zhu, Y., Lin, Z. & Huang, Z. Cryo-EM structure of the type III-E RISPR-Cas effector gRAMP in complex with TPR-CHAT. Cell Res. 32, 1128-1131 (2022).
8. Hemant N Goswami. et al. Molecular mechanism of active Cas7-11 in processing CRISPR RNA and interfering target RNA. Elife 11, e81678 (2022).
9. Yu, G. et al. Structure and function of a bacterial type III-E CRISPR-Cas7-11 complex. Nat. Microbiol. 7, 2078-2088 (2022).
10. Strecker, J. et al. RNA-activated protein cleavage with a CRISPR-associated endopeptidase. Science 378, 874-881 (2022).
11. Kato, K. et al. RNA-triggered protein cleavage and cell growth arrest by the type III-E CRISPR nuclease-protease. Science 378, 882-889 (2022).
12. Liu, X. et al. Target RNA activates the protease activity of Craspase to confer antiviral defense. Mol. Cell 82, 4503-4518 (2022)
13. Babatunde Ekundayo. et al. Structural insights into the regulation of Cas7-11 by TPR-CHAT. Nat Struct Mol Biol. 10.1038/s41594-022-00894-5 (2022).
14. Cui, N. et al. Structural basis for the non-self RNA-acti-vated protease activity of the type III-ECRISPR nuclease-protease Craspase. Nat. Commun. 10.1038/s41467-022-35275-5 (2022)
15. Wang, X. et al. Target RNA-guided protease activity in type III-E CRISPR-Cas system. Nucleic Acids Res 50, 12913-12923 (2022).
(供稿:江涛研究组)
附件下载: