
方显杨研究组与合作者揭示
翻译调控型T-box核糖开关折叠与识别tRNA耦联的结构与动态机制
T-box核糖开关是一类位于革兰氏阳性细菌mRNA的 5'非翻译区的结构元件,其长度通常在300核苷酸以下,可分为适配体结构域和表达平台结构域。T-box核糖开关通过其适配体结构域识别和结合特定的tRNA并感知其3'末端的氨酰化状态,引发下游RNA元件构象状态的转变进而在翻译水平或转录水平调控下游基因的表达。与其它核糖开关一般通过识别小分子代谢物或离子调控基因表达的机制不同,T-box基因表达调控的功能主要是通过两个高度结构化的RNA(T-box核糖开关和tRNA)之间的相互作用实现的。近年来多种T-box核糖开关与tRNA复合物的三维结构已被解析,但是分子内的和分子间的RNA-RNA相互作用是如何促进T-box核糖开关的折叠和结构转变进而形成特定构象状态以发挥生理功能的机制仍不清楚。
2023年11月15日,中国科学院生物物理研究所方显杨研究组与清华大学生命科学学院陈春来研究组合作,在《Nature Communications》在线发表了题为"Structural and dynamic mechanisms for coupled folding and tRNA recognition of a translational T-box riboswitch"的研究论文。在该项研究中,研究人员通过整合应用小角X射线散射(SAXS)技术、单分子荧光共振能量转移(smFRET)技术以及分子动力学模拟,探究了来自皮疽诺卡氏菌(Nocardia farcinica)的ileS T-box核糖开关适配体结构域(包含stem I, stem II和stem IIA/B茎环元件)在溶液中的折叠与构象动态,构建了由镁离子及tRNA介导的T-box核糖开关适配体结构域的折叠自由能面,为深入理解RNA-RNA相互作用对RNA折叠与识别的重要影响以及发展和理性设计靶向RNA的新型抗生素药物提供了理论依据。
在该项研究中,为实现对T-box核糖开关适配体结构域的高密度荧光探针标记(dense labeling)进而利用smFRET技术研究其构象动态,方显杨研究组发展了基于非天然碱基系统(NaM-TPT3)的长链RNA位点特异性荧光探针正交标记技术(图1),通过重叠延伸PCR及体外转录向RNA的特定位点引入化学修饰的非天然碱基,通过点击化学以及NHS酯基与氨基的化学反应将荧光探针与非天然碱基相耦联,从而实现对RNA的位点特异性标记。该方法突破了传统RNA位点特异性标记技术(如化学合成等)在标记长度、标记效率及对结构的干扰等方面的局限性,将极大的推动和拓展smFRET技术在研究长链RNA构象动态以及解析三维溶液结构模型中的应用。
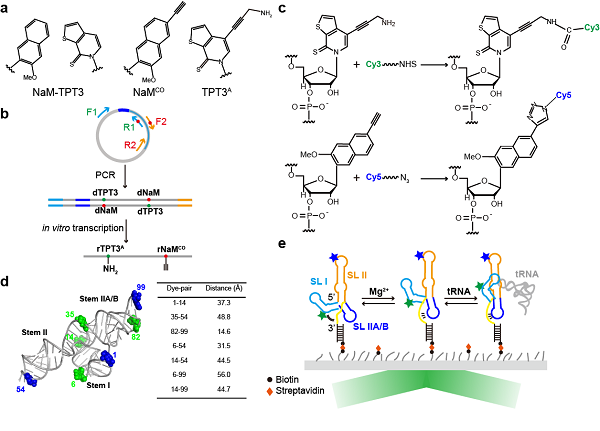
图1 基于非天然碱基系统的RNA位点特异性荧光标记技术
利用基于非天然碱基系统的位点特异性正交荧光标记策略,研究者在T-box核糖开关适配体结构域上选择了七对标记位点,构建了一个密集的标记网络,得以全面的探究tRNA及镁离子诱导的T-box核糖开关适配体的构象动态。研究发现,镁离子可以稳定stem IIA/B假结结构,使其与stem II结构域共轴堆积,进而诱导stem I与stem II结构域的预对接,使T-box折叠为可以结合tRNA的活性构象,而tRNA的结合则进一步诱导了stem I与stem II结构域的对接。此外,研究者也利用SAXS技术结合分子动力学模拟建立了T-box核糖开关不同长度的转录中间产物在不同镁离子条件下的溶液结构模型,并结合突变实验进一步证实了stem IIA/B假结结构以及S-turn结构基序在T-box识别tRNA过程中的重要作用。
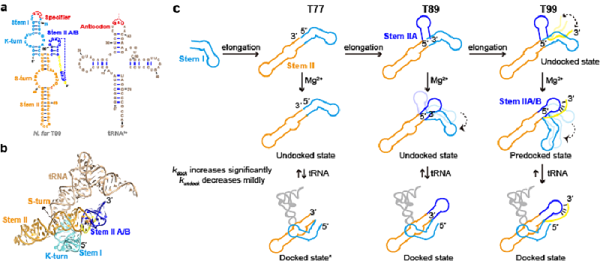
图2 镁离子及tRNA介导的T-box的折叠模型
综合上述实验结果,研究者们提出了镁离子及tRNA介导的T-box核糖开关适配体的共转录折叠模型(图2),揭示了在转录过程中T-box核糖开关各个结构元件相互协同调控促进其折叠,最终保证其能高效、快速识别tRNA的分子机制。总的来说,该项工作揭示了镁离子以及分子内或分子间的RNA-RNA相互作用之间的微妙平衡在调控T-box核糖开关折叠以及功能中的作用。
中国科学院生物物理研究所方显杨研究员与清华大学生命科学学院陈春来副教授为该论文的共同通讯作者。清华大学生命科学学院博士后牛晓林为该论文的第一作者。清华大学生命科学学院2017级博士生徐钟河、中国科学院生物物理研究所研究助理张宇帆参与了该研究工作。美国阿贡国家实验室左孝兵博士为SAXS数据的收集做出了重要贡献。该研究受到国家自然科学基金委、北京市生物结构前沿研究中心、中国科学院先导项目、中国博士后科学基金以及清华大学"水木学者"计划的经费支持。
文章链接:
https://doi.org/10.1038/s41467-023-43232-z
(供稿:方显杨研究组)
附件下载: