
朱冰课题组揭示DNA甲基化维持的动态过程和调控机制及其对衰老和肿瘤发生过程中甲基化选择性丢失的贡献
2020年6月24日,《Cell Research》在线发表了中国科学院生物物理研究所朱冰实验室的研究论文"Kinetics and mechanisms of mitotic inheritance of DNA methylation and their roles in aging-associated methylome deterioration"。该工作解析了细胞有丝分裂中DNA甲基化维持的动态过程和相关调控机制,并发现DNA甲基化维持效率在不同的特征序列环境上有着微小的差异。因此在衰老和肿瘤发生过程中会因为细胞分裂次数的累积而导致特定区域甲基化的选择性丢失。
DNA甲基化作为一种重要的表观修饰,在调控基因的时空特异性表达中扮演着关键的角色,参与了X染色体失活、基因组印记和重复序列抑制等诸多生命过程。哺乳动物细胞的DNA甲基化主要发生在CpG二核苷酸对的C(胞嘧啶)上,并在有丝分裂过程中得以相对稳定的维持,这对细胞保持谱系特性有着重要的意义。然而,由于方法和技术的限制,DNA甲基化维持的效率、动态过程及其相关调控因素研究得并不清楚。
这项工作首先建立了研究DNA甲基化动态维持过程的新方法Hammer-seq。此方法结合了EdU(胸腺嘧啶类似物)标记新合成DNA链、点击化学反应在EdU位点添加biotin标签、Streptavidin免疫沉淀以及Hairpin亚硫酸盐全基因组甲基化测序等技术。Hammer-seq可以测定单个DNA分子内新旧CpG的甲基化状态,从而不仅可以测定甲基化的维持速率,还可以测定维持中可能伴随发生的从头甲基化事件。
研究发现,DNA甲基化可以在复制叉经过后数分钟内迅速恢复到较高的水平,说明存在一个复制偶联的快速维持阶段;而另一方面,即使复制叉经过后数小时,甲基化维持仍然在进行,说明同时存在一个复制非偶联的缓慢维持阶段。
结合Hammer-seq和系统性地突变DNA甲基化维持可能相关的各种蛋白因子,本工作解析了调控复制偶联和非偶联阶段甲基化维持的相关机制。其中,DNMT-PCNA以及UHRF1-LIG1的相互作用促进了复制偶联阶段甲基化的快速维持,UHRF1-H3K9me2/3的相互作用促进了复制非偶联阶段的甲基化维持速率,核小体的存在本质上则会减缓复制非偶联阶段的甲基化维持速率,而LSH相对特异地促进复制晚期区域的复制非偶联阶段的甲基化维持速率。同时,当复制偶联阶段的甲基化维持受到了破坏,甲基化可以在复制非偶联阶段得到很大程度的维持,说明复制非偶联阶段保留了很强的甲基化维持能力。另外,甲基化CpG密度高的区域,不仅具有较高的甲基化维持速率,其从头甲基化发生频率也较高。
衰老和肿瘤发生中均会发生大规模的甲基化丢失,但机制不明。结合分析Hammer-seq的数据,我们发现,衰老和肿瘤发生中甲基化丢失的CpG位点表现出较低的甲基化维持速率和从头甲基化频率。这一结果提示,甲基化维持系统内在的不完美,可能造成一些不易维持的CpG位点在不断的有丝分裂中逐渐丢失甲基化,这或许是衰老和肿瘤中甲基化丢失的重要的机制。
本研究主要在中国科学院生物物理研究所完成,朱冰研究员为通讯作者,明轩博士和张珠强研究员为共同第一作者,朱冰实验室的邹卓宁、董强、贺琦翔、易旸洋和李颖峰等对本工作有重要的贡献。中国科学院生态环境研究中心的汪海林研究员及其博士生吕聪负责了甲基化水平测定的质谱部分。本研究得到了国家自然科学基金委、科技部和中国科学院经费的资助。
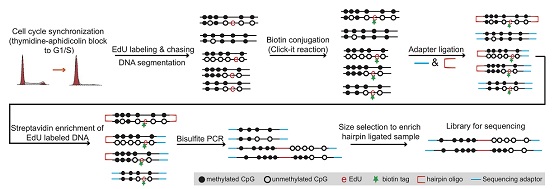
图1:检测DNA甲基化维持水平的新方法Hammer-seq的流程图
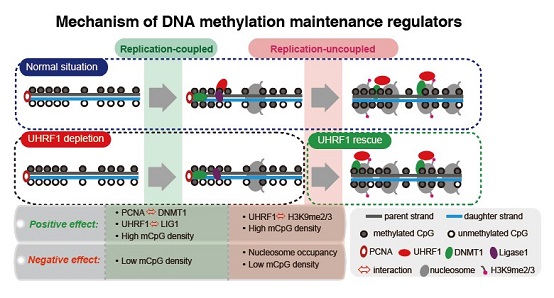
图2:影响DNA甲基化维持速率的各因素的调节机制
文章链接:https://www.nature.com/articles/s41422-020-0359-9
(供稿:朱冰研究组)
附件下载: