
薛愿超课题组开发RIC-seq新技术
2020年5月6日,Nature杂志在线发表了题为“RIC-seq for global in situ profiling of RNA–RNA spatial interactions”的研究论文。该工作开发了能够捕获细胞内RNA原位高级结构及分子间相互作用位点的RIC-seq新技术,解析了HeLa细胞中mRNA和非编码RNA的构象和组织规律,绘制了全基因组增强子-启动子RNA调控网络图谱,并阐明了增强子RNA激活癌基因MYC转录的新机制。
RNA结构在遗传信息传递过程中发挥了关键的调节作用,比如:剪接体中snRNA结构的动态变化保证了剪接的高效和精准进行;核糖体RNA的高级结构对于执行翻译功能是必须的;而端粒RNA组分TERC的假节结构确保了端粒酶的活性和染色体的稳定性。在细胞内,绝大多数RNA通过分子内的碱基配对可形成二级结构,并在RNA结合蛋白的介导下折叠成复杂的三级结构,高度结构化的RNA进而通过与其他RNA分子相互作用以发挥生物学调控功能,因此解析细胞内RNA的原位高级结构及作用靶标是研究其功能机制的关键。虽然目前已有多种研究RNA二级结构和分子间相互作用的高通量测序技术,但是这些实验方法不能有效地捕获RNA的高级结构,而且由于采用体外近端连接,导致假阳性率普遍过高。
针对以上问题,在该项工作中,研究人员开发了RIC-seq新技术,并利用该技术首次绘制了HeLa细胞内RNA-RNA的原位三维作用图谱,揭示了蛋白质编码mRNA以及非编码RNA的空间组织规律与特征。在应用上,该工作率先发现了增强子和启动子非编码RNA之间的相互作用可用于推断其调控网络,并详细解析了超级增强子长链非编码RNA CCAT1-5L与RNA结合蛋白hnRNPK、MYC启动子和增强子RNA结合以改变染色质构象,进而调节癌基因MYC转录的新机制。研究发现RIC-seq技术不仅能检测RNA的高级结构,而且可准确鉴定各类非编码RNA的作用靶标,这为后续深入研究非编码RNA所携带的“结构密码”及其功能性提供了全新的实验工具。此外,该技术在病毒RNA的结构和靶标研究方面也有广阔的应用前景。
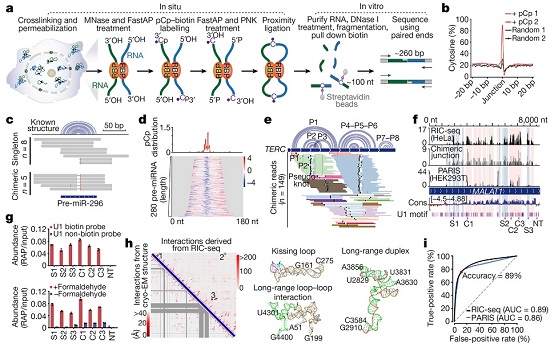
图 RIC-seq可准确捕获RNA的原位高级结构与作用靶标。
该研究工作由中国科学院生物物理研究所、河南师范大学和信阳师范学院合作完成。中国科学院生物物理研究所博士生蔡兆奎、副研究员曹唱唱和吉蕾为本文共同第一作者,薛愿超研究员为通讯作者。该项研究工作获得了国家自然科学基金委非编码RNA重大研究计划、国家重点研发计划和中国科学院先导项目等基金的支持。此外,生物物理研究所蛋白质科学平台和遗传与发育生物学研究所成像平台为本研究提供了设备和技术支持。
文章链接:https://doi.org/10.1038/s41586-020-2249-1
(供稿:薛愿超研究组)
附件下载: