
生物物理所合作研究揭示
生命活动的中心法则是由遗传物质DNA转录生成信使RNA,再由信使RNA翻译成蛋白质,从而完成新陈代谢、生长发育等各项生理功能。然而,细胞(尤其是高等生物细胞)内还存在着大量不翻译成蛋白质的RNA,被称为非编码RNA。它们在基因表达调控等关键生命活动过程中发挥重要作用,与细胞分化、个体发育以及疾病发生与发展密切相关。其中一类功能重要的小非编码RNA是microRNA(miRNA),它们长约21~24个碱基,普遍存在于从线虫到人类等高等生物中,而且很多miRNA的生物学功能在进化过程中保守。自1993年首次发现以来,已有数以万计的miRNA被鉴定,其中一些被作为癌症等疾病的诊断标志物和药物研发靶点。因此,miRNA的功能及其自身的生成与调控机制一直都是生物医学研究热点。
具有功能的miRNA是由一条包含一个颈环结构的更长转录本(又称pri-miRNA)经过两步切割反应而产生。第一步切割反应在细胞核内进行,由Drosha/DGCR8复合物催化完成,其中Drosha为III型RNA切割酶,是核心催化组分,DGCR8为双链RNA结合蛋白,负责招募pri-miRNA底物。核内切割产生长度约60-70个碱基左右的前体miRNA,然后前体miRNA出核,在细胞质由Dicer RNA酶完成第二步切割。第一步在核内的切割反应尤为重要,一方面去除冗长的无关序列,从上千碱基长度的pri-miRNA产生仅60-70个碱基的前体miRNA;另一方面,切割产生的3’端就是最终成熟miRNA的末端,对于miRNA的功能至关重要,因此要求切割位点非常精确。
Drosha/DGCR8复合物作为细胞核内唯一pri-miRNA切割酶是2003-2004年发现的,尽管过去十余年的大量的研究,包括pri-miRNA上的关键序列的鉴定和蛋白质重要功能结构域的分析等,但是pri-miRNA如何被准确识别以及Drosha/DGCR8如何界定切割位点这些重要科学问题一直没有得到清晰的回答。2020年3月27日,《Molecular Cell》杂志在线发表了生物物理所许瑞明课题组与清华大学王宏伟课题组合作完成的题为“Structural Basis For pri-miRNA Recognition by Drosha”的研究论文,利用单颗粒冷冻电镜方法解析了Drosha/DGCR8与pri-miRNA的复合物结构,揭示了pri-miRNA核内加工的分子机制。他们的研究结果证实了Drosha在切割位点界定中的决定性作用,发现Drosha的PAZ、MB helix和dsRBD结构域在pri-miRNA识别和协同完成切割位点定位中发挥重要作用。其中,PAZ结构域在RNA结合前后出现了明显的构象变化,它与MB helix一起,结合在pri-miRNA的单、双链交界区两侧,形成了pri-miRNA关键特性识别的独特模式。其中PAZ结构域与RNA的结合方式完全不同于之前发现的其他蛋白PAZ结构域仅仅识别RNA的3’末端的方式。概括而言,该项研究首先揭示了Drosha特异性识别pri-miRNA的关键序列和结构特性的分子机制,发现了新颖的PAZ结构域构象及其结合RNA的新模式,阐明了困扰研究领域多年的关键分子机制。
此外,这项研究还提出了Drosha/DGCR8复合物存在不同的活性状态。当没有底物RNA时,Drosha的PAZ发卡状双螺旋占据了切割活性中心区域,阻碍Drosha与RNA的结合;而当识别pri-miRNA底物时,该螺旋发生构象变化,促进并稳定了底物的结合。研究人员认为,这是一种从自抑制状态到活化状态的转变,表明Drosha存在活性自调控机制,有益于在体内环境中识别正确底物进行切割。
中国科学院生物物理研究所许瑞明研究员与清华大学生命科学院王宏伟教授为该论文的共同通讯作者,生物物理所金文星副研究员和清华大学王家博士为共同第一作者,工作的主要参与者还有生物物理所的刘超培副研究员。生物物理所章新政研究员、曹端方博士为研究工作提供了有价值的建议。该研究得到了生物物理所生物成像中心和清华大学冷冻电镜和高性能计算平台的大力支持,以及国家自然科学基金、科技部国家重点研发计划,北京市自然科学基金以及中科院青年创新促进会资助。
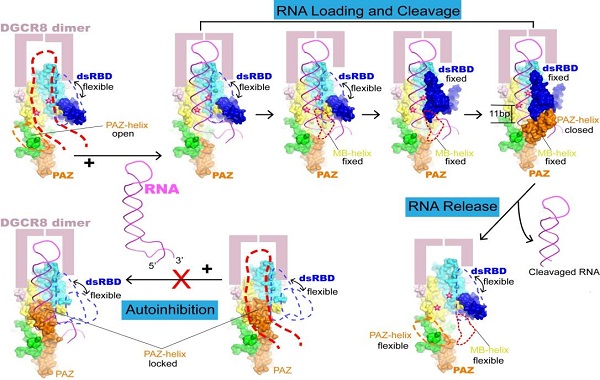
图注:Drosha/DGCR8复合物对底物pri-miRNA的特异性识别及精确切割的机制模型
文章链接:http://www.sciencedirect.com/science/article/pii/S1097276520301441
(供稿:许瑞明研究组)
附件下载: