中国科学院生物物理研究所,表观遗传调控与干预重点实验室,研究组长 中国科学院大学岗位教师
1992 - 1999 湖南师范大学,学士,硕士
1999 - 2003 中国科学院上海生物化学与细胞生物学研究所,博士
2003 - 2011 美国国立卫生研究院癌症研究所(NIH, NCI), 博士后
2011 - 中国科学院生物物理研究所,研究员
染色质结构的动态变化是表观遗传调控的重要基础,与DNA复制、基因转录、DNA损伤修复、基因组稳定性维持等重要生物学事件密切相关。染色质动态源于DNA或组蛋白的序列及化学性质的改变,以及核小体组成和染色质结构的差异,同时也受到组蛋白变体、组蛋白伴侣、组蛋白修饰酶、染色质重塑复合物、非编码RNA等多种表观因子的调节。
本课题组关注的科学问题是染色质动态的调控及功能,我们将运用包括冷冻电镜在内的多种结构生物学手段,结合生物化学、生物物理学、酵母遗传学、单分子力谱、计算生物学等方法,开展两个方向的研究:
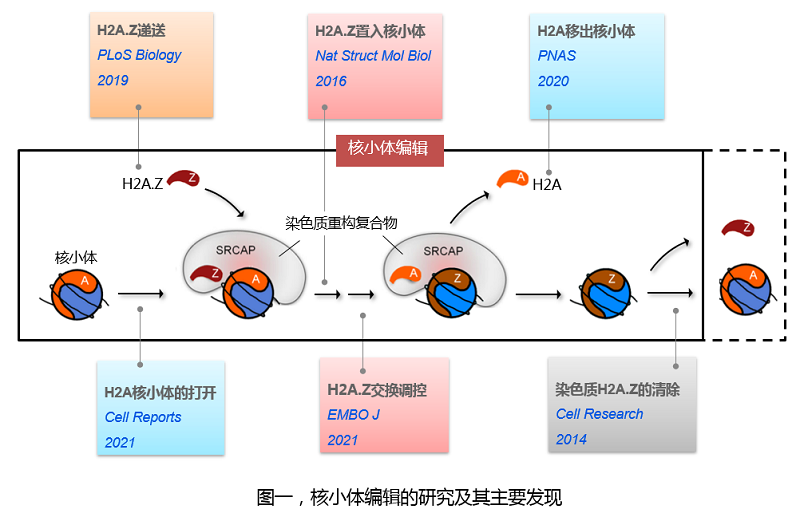
1. 核小体编辑
研究染色质重构复合物如何催化组蛋白变体对常规组蛋白进行替换,以及如何实现核小体编辑的人工操控。核小体编辑通过将组蛋白H2A变体精准定位到基因组特定区域进行基因表达调控,其功能异常与癌症等多种疾病密切相关。阐明核小体编辑的作用机制有助于新型表观遗传编辑技术的研发(图一)。
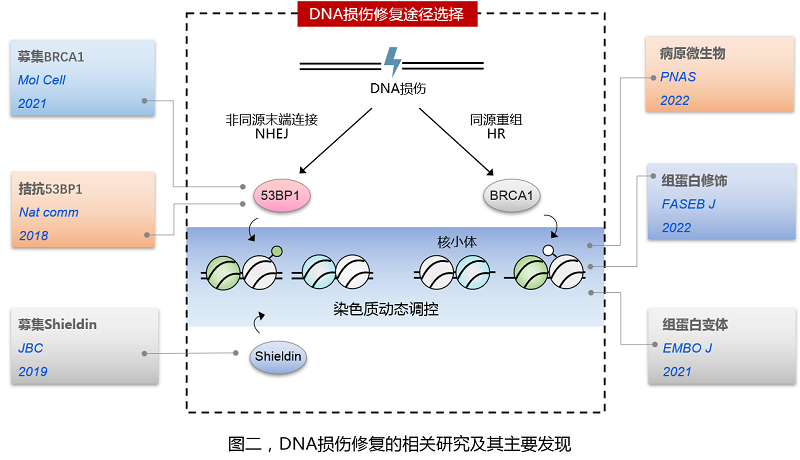
2. DNA损伤修复调控
研究细胞如何选择特定的修复途径对DNA损伤进行修复。DNA损伤"合成致死"效应在癌症治疗中的作用,催生了PARP抑制剂等一线抗癌药物,阐明DNA损伤修复的调控机制有助于发现新的药物靶点,促进抗癌药物的研发(图二)。
发表论文
1. Huang L, Wang Y, Long H, Zhu H, Wen Z, Zhang L, Zhang W, Guo Z, Wang L, Tang F, Hu J, Bao K, Zhu P, Li G, Zhou Z. (2023) Structural insight into H4K20 methylation on H2A.Z-nucleosome by SUV420H1. Mol Cell. 83(16):2884-2895. OPEN
2. Chen J, Lu Z, Gong W, Xiao X, Feng X, Li W, Shan S, Xu D, Zhou Z. (2022) Epstein-Barr virus protein BKRF4 restricts nucleosome assembly to suppress host antiviral response. Proc Natl Acad Sci USA.119 (37): e2203782119. OPEN
3. Shi L, Huang L, Long H, Song A, Zhou Z. (2022) Structural basis of nucleosomal H4K20 methylation by methyltransferase SET8. FASEB J. 36(6):e22338. OPEN
4. Dai L, Dai Y, Han J, Huang Y, Wang L, Huang J, Zhou Z. (2021) Structural insight into BRCA1-BARD1 complex recruitment to damaged chromatin. Mol Cell. 81(13): 2765-2777. OPEN
5. Dai L, Xiao X, Pan L, Shi L, Xu N, Zhang Z, Feng X, Ma L, Dou S, Wang P, Zhu B, Li W, Zhou Z. (2021) Recognition of the inherently unstable H2A nucleosome by Swc2 is a major determinant for unidirectional H2A.Z exchange. Cell Rep. 35(8):109183. OPEN
6. Zhou N, Shi L, Shan S, Zhou Z. (2021) Molecular basis for the selective recognition and ubiquitination of centromeric histone H3 by yeast E3 ligase Psh1. J Genetics Genomics. 48(6):463-472. OPEN
7. Zhou M, Dai L, Li C, Shi L, Huang Y, Guo Z, Wu F, Zhu P, Zhou Z. (2021) Structural basis of nucleosome dynamics modulation by histone variants H2A.B and H2A.Z.2.2, EMBO J. 40(1): e105907. OPEN
8. Huang Y, Sun L, Pierrakeas L, Dai L, Pan L, Luk E, Zhou Z. (2020) Role of a DEF/Y motif in histone H2A-H2B recognition and nucleosome editing. Proc Natl Acad Sci USA. 117(7): 3543–3550. OPEN
9. Wang Y, Liu S, Sun L, Xu N, Shan S, Wu F, Liang X, Huang Y, Luk E, Wu, C, Zhou Z. (2019) Structural insights into histone chaperone Chz1-mediated H2A.Z recognition and histone replacement. PLoS Biol. 17(5): e3000277. OPEN
10. Dai Y, Zhang A, Shan S, Gong Z, Zhou Z.(2018) Structural basis for recognition of53BP1 tandem Tudor domain by TIRR. Nat Commun. 9(1):2123. OPEN
11. Liang X, Shan S, Pan L, Zhao J,Ranjan A, Wang F, Zhang Z, Huang Y, Feng H, Wei D, Huang L, Liu X, Zhong Q, Lou J, Li G, Wu C, Zhou Z. (2016) Structural basis of H2A.Z recognition by SRCAP chromatin-remodeling subunit YL1. Nat Struct Mol Biol. 23(4):317-325. OPEN
12. Mao Z, Pan L, Wang W, Sun J, Shan S, Dong Q, Liang X, Dai L, Ding X, Chen S, Zhang Z, Zhu B, Zhou Z. (2014) Anp32e, a higher eukaryotic histone chaperone directs preferential recognition for H2A.Z. Cell Res. 24(4):389–399. OPEN
Invited Review
1. Xiao S, Wang Y, Shan S, Zhou Z. (2023) The interplay between viral molecular mimicry and host chromatin dynamics. Nucleus. 14(1): 2216560. OPEN
2. Huang Y, Dai Y, Zhou Z. (2020) Mechanistic and structural insights into histone H2A-H2B chaperone in chromatin regulation. Biochem J. 477 (17): 3367-3386. (review) OPEN
3. Huang Y, Zhou Z. (2018) Recent progress in histone chaperones associated with H2A-H2B type histones. Prog Biochem Biophys. 45(9): 971-980. (review) OPEN
4. Zhou J, Feng X, Zhou Z. (2015) Chromatin assembly of histone variants. Prog Biochem Biophys. 42(11):1003~1008. (review) OPEN
(资料来源:周政研究员,2024-09-14)
-
 单珊副研究员,中国科学院青促会会员,2012年博士毕业于清华大学后加入实验室,主要研究方向为DNA损伤修复。
单珊副研究员,中国科学院青促会会员,2012年博士毕业于清华大学后加入实验室,主要研究方向为DNA损伤修复。 -
 冯晓利博士,工程师,2009年毕业于郑州大学,2011年加入实验室。
冯晓利博士,工程师,2009年毕业于郑州大学,2011年加入实验室。 -
 黄莉博士,副研究员,2020年博士毕业于中国科学院生物物理研究所,2012年加入实验室。
黄莉博士,副研究员,2020年博士毕业于中国科学院生物物理研究所,2012年加入实验室。 -
 戴霖昌博士,副研究员,中国科学院青年促进会会员,2018年博士毕业于中国科学技术大学,2012年加入实验室。
戴霖昌博士,副研究员,中国科学院青年促进会会员,2018年博士毕业于中国科学技术大学,2012年加入实验室。
-
 朱浩强本科毕业于华南师范大学,2017年加入实验室,硕博连读生。
朱浩强本科毕业于华南师范大学,2017年加入实验室,硕博连读生。 -
 阳鑫本科毕业于湖南农业大学,2020年加入实验室,硕博连读生。
阳鑫本科毕业于湖南农业大学,2020年加入实验室,硕博连读生。 -
 唐方毅本科毕业于西北农林科技大学,2020年加入实验室,硕博连读生。
唐方毅本科毕业于西北农林科技大学,2020年加入实验室,硕博连读生。 -
 黄文康本科毕业于湖南大学,硕士研究生毕业于兰州大学,2021年加入实验室,科研博士生。
黄文康本科毕业于湖南大学,硕士研究生毕业于兰州大学,2021年加入实验室,科研博士生。 -
 肖淑敏本科毕业于郑州大学,2021年加入实验室,硕博连读生。
肖淑敏本科毕业于郑州大学,2021年加入实验室,硕博连读生。 -
 王亚静本科毕业于吉林大学,2021年加入实验室,硕博连读生。
王亚静本科毕业于吉林大学,2021年加入实验室,硕博连读生。 -
 周浩天本科毕业于中南民族大学,2022年加入实验室,硕士生。
周浩天本科毕业于中南民族大学,2022年加入实验室,硕士生。 -
 马建明本科毕业于华中科技大学,2022年加入实验室,硕博连读生。
马建明本科毕业于华中科技大学,2022年加入实验室,硕博连读生。 -
 肖之奇本科毕业于安徽农业大学,2022年加入实验室,硕士研究生。
肖之奇本科毕业于安徽农业大学,2022年加入实验室,硕士研究生。 -
 靳红叶本科毕业于南开大学,2023年加入实验室,硕士研究生。
靳红叶本科毕业于南开大学,2023年加入实验室,硕士研究生。 -
 付淑笙本科毕业于中国农业大学,2023年入组,硕士研究生。
付淑笙本科毕业于中国农业大学,2023年入组,硕士研究生。 -
 李玉萍本科毕业于西南大学,2024年加入实验室,硕士研究生。
李玉萍本科毕业于西南大学,2024年加入实验室,硕士研究生。
-
 梁小平博士,2016年离组,Johns Hopkins University, US
梁小平博士,2016年离组,Johns Hopkins University, US -
 徐宁博士,2017年离组,清华大学
徐宁博士,2017年离组,清华大学 -
 王云云博士,2018年离组,药明生物
王云云博士,2018年离组,药明生物 -
 谢晓燕博士,2019年离组,Van Andel Institute, US
谢晓燕博士,2019年离组,Van Andel Institute, US -
 周驹俊博士,2019年离组,MD Anderson Cancer Center, US
周驹俊博士,2019年离组,MD Anderson Cancer Center, US -
 吴飞博士,2019年离组,Laboratory of Molecular Biology, UK
吴飞博士,2019年离组,Laboratory of Molecular Biology, UK -
 潘露博士,2020年离组, 中国军事医学科学院
潘露博士,2020年离组, 中国军事医学科学院 -
 戴亚鑫博士,2021年离组,St.Jude Children's Research Hospital, US
戴亚鑫博士,2021年离组,St.Jude Children's Research Hospital, US -
 黄艳博士,2021年离组,St.Jude Children's Research Hospital, US
黄艳博士,2021年离组,St.Jude Children's Research Hospital, US -
 谌娇博士,2021年离组,信达生物
谌娇博士,2021年离组,信达生物 -
 周宁博士,2024年离组,University of Texas Southwestern Medical Center, US
周宁博士,2024年离组,University of Texas Southwestern Medical Center, US -
 史刘歆博士,2022年离组,华大基因
史刘歆博士,2022年离组,华大基因 -
 周英博博士,2023年离组,药明生物
周英博博士,2023年离组,药明生物 -
 王龙歌博士,2024年离组,济南市属医疗机构
王龙歌博士,2024年离组,济南市属医疗机构