

中国科学院青年创新促进会会员 北京市科技新星计划入选者
1997.07 - 2001.09 南昌大学 生物科学工程系 学士
2001.08 - 2004.07 吉林大学(农学部)生物化学与分子生物学教研室 硕士
2004.08 - 2007.06 中国协和医科大学 基础所 生物化学与分子生物学系 博士
2007.07 - 2010.08 中国科学院生物物理研究所 助理研究员
2010.09 - 2013.11 中国科学院生物物理研究所 副研究员
2015.09 - 2016.09 美国UCLA微生物免疫分子遗传系 高级访问学者
2013.12 - 2017 中国科学院生物物理研究所 研究员
(2013年12月获中国科学院生物物理研究所“青年研究员计划”支持)
2014年 北京市科技新星计划入选者
2013年 荣获中科院“卢嘉锡青年人才奖”
国际基金项目评审人(如捷克科学基金会(Czech Science Foundation)项目评审人)和学术论文评审人(如EMBO J,PNAS,Acta Crystallographica Section D,Trends in Microbiology,Protein & Cell,J Gen Virol,J Struct Biol,Science China: Life Science,生物化学与生物物理进展,科学通报,Current Chemical Biology等)。
运用X射线晶体学、冷冻电镜和其它生物物理技术研究病原体感染和宿主天然免疫反应的分子机制。
真核生物的天然免疫(又称固有免疫或非特异性免疫)反应(innate immune response),发生于系统发育的早期和抗感染应答的初期阶段,是机体抵抗病原微生物入侵、保护自身的第一道屏障。天然免疫系统一方面可以通过炎症反应等机制直接清除感染,另一方面还可以诱导获得性免疫(acquired immunity)系统的活化,进而激发更加高效和特异性的免疫应答,故天然免疫是免疫之本。天然免疫系统通过机体的模式识别受体(pattern-recognition receptors,PRRs)对入侵微生物中高度保守的“分子标签”(molecular signature)——病原相关分子模式(pathogen-associated molecular patterns,PAMPs)进行特异性识别,从而感知细菌、病毒等外来微生物入侵并激活不同的胞内信号通路和基因表达,启动机体免疫反应。天然免疫如同机体的眼睛,监视与识别各种不同的疾病相关分子模式。因此,了解机体免疫系统对核酸识别和检测的分子基础是天然免疫研究的重要内容。
课题组自2008年起从事天然免疫反应信号通路的结构与功能研究,已取得了一系列研究成果。病原微生物引发天然免疫反应尤其是STING介导的信号通路引起I型IFN产生的研究正如火如荼,仍有许多科学问题亟待解决。课题组综合运用X-射线晶体学方法,NMR,Cryo-EM和细胞生物学技术致力于阐明胞内DNA感受因子识别、DNA结合、激活STING引起I型IFN产生过程中关键科学问题。
1. 2016年11月1日《PNAS》杂志在线发表了生物物理所题为“Structural basis for DNA recognition by STAT6”的研究论文,首次揭示STAT6对N4位点DNA的识别机制。
STING有关的信号通路一直是我们关注的领域,STAT6因与STING有相互作用引起我们的注意。此前报道STAT6作为信号转导及转录激活因子家族中的一员,参与到JAK-STAT信号通路,受到IL-4/IL-13的调控,将信号从细胞膜传递到细胞核并激活下游一系列基因的转录表达,在Th2细胞的分化过程中起到关键作用。STAT6表达与炎症和多种肿瘤关系密切,可能成为新的肿瘤标志物,检测STAT6分子具有重要临床意义,可以作为判断预后的指标和治疗的新靶点,筛选出该通路的抑制分子就可能成为STAT6通路的异常所致疾病的潜在预防和治疗的作用药物。STAT6家族蛋白作为转录因子首先在胞质中磷酸化后形成二聚体,二聚体的STAT6进入细胞核并识别和结合靶基因启动子区域启动一系列基因表达。STAT家族蛋白识别相似的DNA序列,其基本结构为TTCN3/4GAA,但是不同的STAT蛋白所识别的DNA回文序列的中间间隔区长度不同。在哺乳动物STAT家族中,STAT6是唯一一个主要识别N4位点启动子(TTCN4GAA)的家族成员,而其他STAT家族蛋白以识别N3位点启动子(TTCN3GAA)为主。对于这种对DNA识别差异的结构基础,至今仍缺乏比较合理的机制阐述,尤其STAT6对N4位点DNA的识别机制是过去10多年一直没解决的一个重要科学问题。
我们采用结构生物学方法解析了STAT6核心区的磷酸化二聚体晶体结构,同时完成其与N4位点DNA和N3位点DNA两个复合物三维精细结构的解析工作,这是其它STAT家族蛋白中从来没有过的,因此我们有足够的结构资源进行结构分析。STAT6核心区的磷酸化二聚体结构与其它已解析STAT蛋白结构采用相似机制形成“V”字形。通过结构比对发现,结合核酸以后,STAT6二聚体发生一个明显的旋转型的构象变化,同时发现结合N4位点DNA和N3位点DNA后STAT6关键氨基酸H415在DNA双螺旋轴方向的位置变化距离与DNA双螺旋中相邻2个碱基之间的垂直距离(approximately 3.4?rise/bp along the axis of a B-DNA double helix)相符。这部分工作是在STAT蛋白家族中首次针对同一STAT蛋白观察到结合DNA前后的构象变化。分子动力学模拟以及小角散射实验的结果都证明了STAT6的构象在DNA结合后处于更稳定的状态。通过与其它已报道STAT蛋白结构比较发现,STAT6磷酸化二聚体中“V”形的夹角比其它STAT蛋白要大一些,导致二聚体结构中的两个分子之间的动态变化范围更广。同时我们发现,STAT6 DNA结合结构域上的H415是唯一与DNA碱基直接相互作用的氨基酸,这引起我们的关注。序列分析发现除STAT5以外,在其它STAT蛋白上H415所对应的氨基酸是N(Asn)。体外实验发现,STAT6的H415突变成N后识别N4位点DNA的能力明显下降而识别N3位点DNA的能力上升,而将STAT1上相应的氨基酸N460突变成H后识别N3位点DNA的能力明显下降而识别N4位点DNA的能力上升。细胞水平实验也证明STAT6的H415对STAT6特异性结合N4位点DNA具有关键作用。STAT5不能广泛识别N4位点DNA可能是因为一方面STAT5二聚体夹角稍小,另一方面H471的侧链伸展方向与STAT6的H415不同。
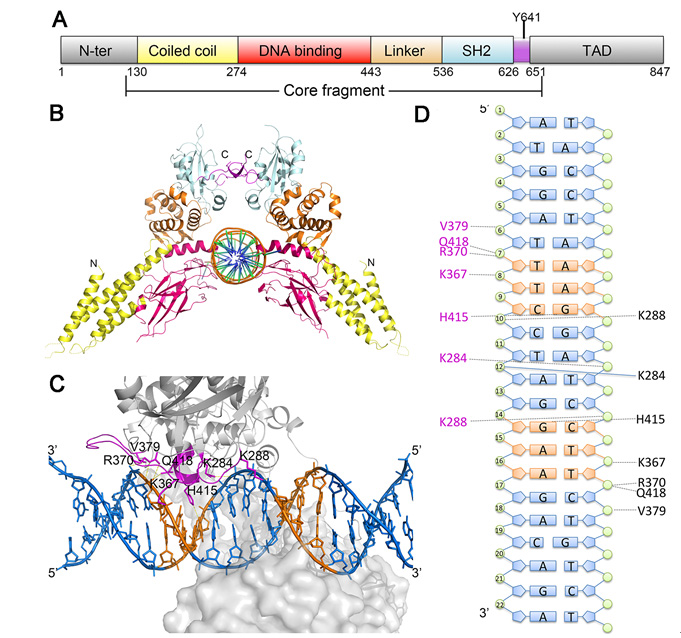
图1:STAT6核心片段在全长中所在的区域及STAT6核心片段与N4位点DNA复合物结构。
(Li et al., PNAS. 2016 Nov 15;113(46):13015-13020)
2. 2013年5月,美国科学院院刊《PNAS》发表了题为 “Structure of the Leanyer Orthobunyavirus Nucleoprotein-RNA complex reveals unique architecture for RNA encapsidation”的研究成果。首次报道了正布尼亚病毒属(Orthobunyavirus genus)的核蛋白(N)和单链RNA复合物的晶体结构,阐述了N蛋白保护病毒基因组RNA的分子机制,为此种属病毒疾病的预防和治疗提供了有力的理论基础。同期配发同行评价(Zheng W,Tao YJ. Genome encapsidation by orthobunyavirus nucleoproteins. Proc Natl Acad Sci U S A. 2013 110 (22) 8769-8770),并以“From the cover”形式发表。
单链负链RNA病毒,是威胁人类健康的重要杀手。高致病性的狂犬病毒,流感病毒等均属于此列。作为最大的负链RNA病毒家族,布尼亚病毒科共包括5个属,超过370多种,引发多种传播疾病。该类病毒基因组包括三条长度不同的RNA单链,依据长度分别命名为L、M、S,相应编译RNA聚合酶、病毒衣壳 糖蛋白Gn/Gc和病毒核蛋白(N)。在此类病毒的转录、复制和增殖过程中,N蛋白会全程结合并保护病毒基因组RNA,避免裸露的RNA被宿主细胞的抗病 毒分子破坏。结合N 蛋白的RNA链作为转录和复制的模板在病毒的生命周期中发挥不可替代的重要作用。正布尼亚病毒属则是该家族最早被发现且种类最多(超过160种)的属,了解其N蛋白是如何结合并保护病毒基因组RNA,是研究并阻击此类病毒疾病危害人类的重要突破口。
课题组从正布尼亚病毒属的代表性病毒Leanyer virus的N蛋白入手,解析了Leanyer-N结合单链RNA的复合物晶体结构。从结构中可以清晰发现,N蛋白由N端和C端两部分结构域组成,在它们中间会形成了强烈的正电荷沟槽,此部位结合并保护单链病毒RNA。同时,在N蛋白的N端和C端分别伸出约20个氨基酸的柔性手臂,分别搭在前后相邻的两个N蛋白上,稳定蛋白的多聚化形式。四聚体内环处形成的连续的正电荷沟槽结合并保护病毒RNA链。体外的负染-冷冻电镜实验结果也清晰表明,随着RNA链长度的增加,N蛋白结合在RNA链上并形成一条线性的不规则病毒RNP。此外,该组还解析了体外重组的N蛋白和单链DNA复合物的晶体结构,类似于N-RNA复合物模型,进一步阐述该N蛋白结合病毒基因组的模式。该成果从分子层 次阐述了正布尼亚病毒基因组的保护机理,为理解其生活模式和预防此类疾病作出重要贡献。
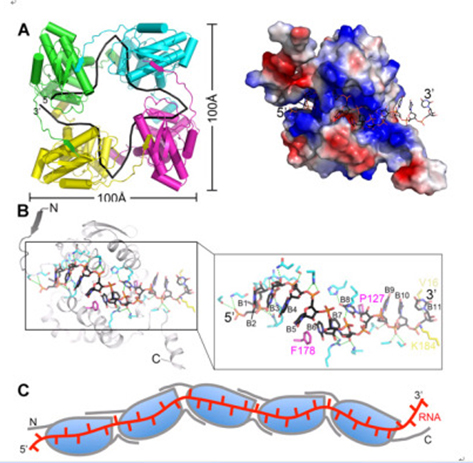
图2:(A) LEAV-N:RNA复合物以及RNA结合的正电荷沟。(B) LEAV-N和RNA相互作用。(C)线性的病毒RNP模型。
(Niu F et al.,PNAS. 2013 May 28;110(22):9054-9.)
3. 2012年5月10日,国际著名免疫学杂志《Immunity》在线发表了重要天然免疫系统信号分子STING结构与功能研究方面的合作研究成果,该论文题为“Structural Analysis of the STING Adaptor Protein Reveals a Hydrophobic Dimer Interface and Mode of Cyclic di-GMP Binding”(DOI:10.1016/j.immuni.2012.03.019)。
天然免疫反应是机体抵抗病原体入侵、保护自身的第一道防线。自2008年以来,关于干扰素刺激因子STING(STimulator of INterferon Genes,又名MITA,ERIS或MPYS)通过引起I型干扰素的产生激发机体天然免疫反应的一系列研究引起了人们的广泛关注。美国迈阿密大学医学院癌症研究中心的Glen N. Barber课题组分别于2008年10月和2009年10月在Nature上报导称,新发现的STING是启动机体天然免疫系统抵抗细菌或病毒感染的重要分子。武汉大学舒红兵院士课题组发现,STING作为天然免疫抗病毒信号接头分子将病毒感知受体和IRF3激活、I型干扰素产生联系起来。后来的研究还发现E3泛素连接酶RNF5与STING相互作用,并负调节病毒触发的下游信号通路。北京大学蒋争凡教授课题组报道了STING引起I型干扰素产生的信号通路激活需要STING二聚化。他们还发现了一条通过STING-TBK1活化转录因子STAT6,从而连接天然免疫与适应性免疫的信号传导通路,为人们进一步认识免疫系统如何防御病原微生物感染的机制提供了新思路。2010年日本学者Akira课题组发表文章称TRIM56与STING相互作用并泛素化STING K150位点。该泛素化修饰诱导STING二聚化,而二聚化又是STING募集TBK1、引起干扰素产生的前提条件。然而,该研究不能解释胞质内的TRIM56蛋白是如何实现跨膜(因为153-173肽段是预测的最后一个跨膜区)泛素化STING K150位点。去年9月,Burdette等人发现,STING在病原菌和病毒感染时角色不同:既是病原菌所分泌的第二信使cyclic di-GMP(c-di-GMP)的感受因子(sensor),又是宿主感知病毒核酸产生I型干扰素反应的信号接头分子(adaptor)。该研究发表在Nature上,引起天然免疫领域学者的广泛关注。总之,近年来有关STING的大量研究报导表明了该分子在天然免疫信号通路中的重要性。同时,不同课题组间某些研究结果的不一致性更加激发了人们对STING进行更深入探讨的热情。
STING蛋白包含一个预测的N端五次(有的文献认为四次)跨膜部分(1-173aa)和C端胞内可溶部分(174-379aa),如图2所示。有关N端的功能目前仍不清楚,但和其在细胞内定位有关。C端胞内结构域(C-terminal domain,CTD)可通过募集相互作用分子而行使重要功能。STING氨基酸序列与PDB中任何已知结构的蛋白质没有同源性,预示它可能拥有独特的三维结构。此外,到目前为止,仍有许多关键科学问题悬而未决,如STING激活TBK1-IRF3信号通路的分子机制,TRIM56如何实现跨膜泛素化STING K150的,STING是如何结合c-di-GMP的?针对以上问题,我们开展了针对STING CTD的结构生物学和细胞生物学研究。
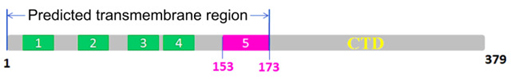
图3:STING全长的二级结构组织模式。它包含N端跨膜区和C端可溶结构域CTD。
针对上述科学问题,本研究综合运用了X-射线晶体学,生物化学和细胞生物学技术。我们先后解析了STING CTD以及STING CTD与c-di-GMP二元复合物的晶体结构。发现STING CTD具有一种独特的构架(Unique architecture),阐明了STING CTD形成功能性二聚体的分子机制,文献报道的最后一个跨膜区(153-173aa)其实并不是跨膜区而是STING CTD形成同源二聚体的疏水相互作用界面。STING CTD与c-di-GMP以一种全新的模式结合。据我们所知该结构是第一个发表的哺乳动物来源的蛋白质与c-di-GMP形成的复合物晶体结构(图3)。此外,我们还发现c-di-GMP能促进STING与TBK1结合,诱导I型干扰素的产生,从而激发机体抵抗病原体入侵的免疫反应。该研究工作的完成有助于深入了解STING在天然免疫信号通路中的作用,为揭示宿主细胞感知病原菌入侵的分子机制提供了直接的结构生物学证据,同时也为设计新的环鸟苷二磷酸类似物疫苗佐剂或免疫治疗药物奠定基础。
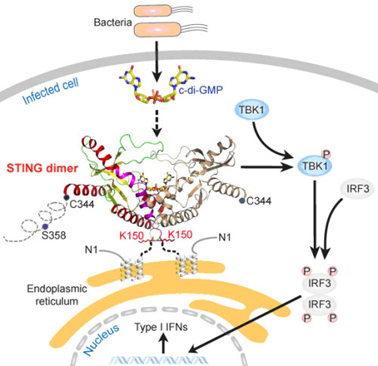
图4:病原菌引起的STING天然免疫信号通路激活示意图
(Ouyang S et al.,Immunity. 2012 Jun 29;36(6):1073-86.)
发表论文
1. Li J#,Rodriguez J#,Niu F,Pu M,Wang J,Hung LW,Shao Q,Zhu Y,Ding W,Liu Y,Da Y,Yao Z,Yang J,Zhao Y,Wei GH,Cheng G,Liu ZJ,Ouyang S*. Structural basis for DNA recognition by STAT6. Proc Natl Acad Sci USA. 2016 Nov 15;113(46):13015-13020. (*通讯作者。This article is a PNAS Direct Submission)
◆ 同行评价:王璞玥,杨正宗,杜生明. STAT6识别N4位点DNA的结构基础被揭示. SCIENTIA SINICA Vitae. DOI: 10.1360/N052016-00324.
Zhou X,Jiang Z. N4 DNA Recognition by STAT6 – Structural and Functional Implications. Protein Cell. 2017 Feb 20. doi: 10.1007/s13238-017-0380-z.
2. Jiang Y,Zhu Y,Liu ZJ, Ouyang S*. The emerging roles of the DDX41 protein in immunity and diseases. Protein Cell. 2017 Feb;8(2):83-89. (*通讯作者)
3. Jiang Y,Zhu Y,Qiu W,Liu YJ,Cheng G,Liu ZJ*,Ouyang S*. Structural and functional analyses of human DDX41 DEAD domain. Protein Cell. 2017 Jan;8(1):72-76. (*通讯作者)
4. Chu Y#,Zhu Y#,Chen Y,Li W,Zhang Z,Liu D,Wang T,Ma J,Deng H,Liu ZJ,Ouyang S*, Huang L*. aKMT Catalyzes Extensive Protein Lysine Methylation in the Hyperthermophilic Archaeon Sulfolobus islandicus but is Dispensable for the Growth of the Organism. Mol Cell Proteomics. 2016 Sep;15(9):2908-23. (*通讯作者)
5. Wang L,Valderramos SG,Wu A,Ouyang S,Li C,Brasil P,Bonaldo M,Coates T,Nielsen-Saines K,Jiang T,Aliyari R,Cheng G. From Mosquitos to Humans: Genetic Evolution of Zika Virus. Cell Host Microbe. 2016 Apr 14. pii: S1931-3128(16)30142-1. (已被引用40次)
6. Jiang Y,Jiang H,Zhou S,Meng B,Liu ZJ*, Ouyang S*. Crystal structure of hGEF-H1 PH domain provides insight into incapability in phosphoinositide binding. Biochem Biophys Res Commun. 2016 Mar 18;471(4):621-7. (*共同通讯作者)
7. Ni X#,Ru H#,Ma F#,Zhao L#,Shaw N,Feng Y,Ding W,Gong W,Wang Q, Ouyang S*, Cheng G*,Liu ZJ*. New insights into the structural basis of DNA recognition by HINa and HINb domain of IFI16. J Mol Cell Biol. 2016 Jan 12;8 (1):51-61. (*共同通讯作者,封面文章)
8. Liu B#,Ouyang S#, Makarova K,Xia Q,Zhu Y,Li Z,Guo L,Koonin E,Liu ZJ*,Huang L*. A primase subunit essential for efficient primer synthesis by an archaeal eukaryotic-type primase. Nat Commun. 2015 Jun 22;6:7300. (#共同第一作者)
9. Zhao L#,Hua T#,Crowley C,Ru H,Ni X,Shaw N,Jiao L,Ding W,Jiang Y,Hung LW,Liu L,Ye K,Ouyang S*, Cheng G*,Liu ZJ*. Structural analysis of asparaginyl endopeptidase reveals the activation mechanism and a reversible intermediate maturation stage. Cell Res. 2014 Mar;24(3):344-58. (*共同通讯作者,已被引用21次)
10. Gu J,Feng Y,Feng X,Sun C,Lei L,Ding W,Niu F,Jiao L,Yang M,Li Y,Liu X,Song J,Cui Z,Han D,Du C,Yang Y, Ouyang S*, Liu ZJ*,Han W*. Structural and biochemical characterization reveals LysGH15 as an unprecedented “EF-hand-like” calcium-binding phage lysin. PLOS Pathogens. 2014 May 15;10(5):e1004109. (*共同通讯作者,已被引用16次)
11. Meng B,Wu D,Gu J, Ouyang S,Ding W,Liu ZJ. Structural and functional analyses of human tryptophan 2,3-dioxygenase. 2014. PROTEINS: Structure,Function,and Bioinformatics. 2014 Nov;82(11):3210-6.
12. Jiao L#,Ouyang S#, Shaw N,Song G,Feng Y,Niu F,Qiu W,Zhu H,Hung LW,Zuo X,Shtykova E,Zhu P,Dong YH,Xu R*,Liu ZJ*. Mechanism of the Rpn13-induced activation of Uch37. Protein Cell. 2014 Aug;5(8):616-30. (#共同第一作者)
13. Zhang L,Mo J,Swanson K,Wen H,Petrucelli A,Gregory S,Zhang Z,Schneider M,Jiang Y,Fitzgerald K, Ouyang S,Liu ZJ,Damania B,Shu HB,Duncan J,Ting J. NLRC3,a member of the NLR family of proteins,is a negative regulator of innate immune signaling induced by the DNA sensor STING. Immunity. 2014 Mar 20;40(3):329-341. (已被引用60次)
◆ 同行评价:Mangan M,Latz E. NLRC3 Puts the Brakes on STING. Immunity. 2014 Mar 20;40(3):305-306.
14. Niu F,Ru H,Ding W,Ouyang S*, Liu ZJ*. Structural biology study of human TNF receptor associated factor 4 TRAF domain. Protein Cell. 2013,4(9):687-694. (*共同通讯作者,封面文章)
15. Niu F#,Shaw N#,Wang Y#,Jiao L#,Ding W,Upurd H, Ouyang S*, Cheng G*,Liu ZJ*. From the Cover: Structure of the Leanyer Orthobunyavirus Nucleoprotein-RNA complex reveals novel architecture for RNA encapsidation. Proc Natl Acad Sci USA. 2013 May 28;110(22):9054-9. (*共同通讯作者,已被引用29次。This article is a PNAS Direct Submission)
◆ 同行评价:Zheng W,Tao YJ. Genome encapsidation by orthobunyavirus nucleoproteins. Proc Natl Acad Sci U S A. 2013 110 (22) 8769-8770.
16. Jiao L#, Ouyang S#, Liang M#,Niu F#,Shaw N,Wu W,Ding W,Jin C,Peng Y,Zhu Y,Zhang F,Wang T,Li C,Zuo X,Luan CH,Li D,Liu ZJ*. Structure of severe fever with thrombocytopenia syndrome virus nucleoprotein in complex with Suramin reveals therapeutic potentials. J Virol. 2013 Jun;87(12):6829-39. (#共同第一作者,已被引用34次)
17. Shaw N,Ouyang S,Liu ZJ. Binding of bacterial secondary messenger molecule c di-GMP is a STING operation. Protein Cell. 2013 Feb;4(2):117-29.
18. Parvatiyar K*,Zhang Z*,Teles RM,Ouyang S,Jiang Y,Iyer SS,Zaver SA,Schenk M,Zeng S,Zhong W,Liu ZJ,Modlin RL,Liu YJ,Cheng G. The helicase DDX41 recognizes the bacterial secondary messengers cyclic di-GMP and cyclic di-AMP to activate a type I interferon immune response. Nat Immunol. 2012 Nov 11;13(8):1155-1161. (封面文章,已被引用达173次)
◆ 同行评价:“News and Views”by Andrew G Bowie:Innate sensing of bacterial cyclic dinucleotides: more than just STING. Nat Immunol. 2012 Dec;13(12):1137-9.
19. Ouyang S#, Song X#,Wang Y#,Ru H#,Shaw N,Jiang Y,Niu F,Zhu Y,Qiu W,Parvatiyar K,Li Y,Zhang R,Cheng G*,Liu ZJ*. Structural Analysis of the STING Adaptor Protein Reveals a Hydrophobic Dimer Interface and Mode of Cyclic di-GMP Binding. Immunity. 2012 Jun;36(6):1073-1086. (#共同第一作者,已被引用达119次) (作为当年具有重要影响力的科研成果被中国科学院《2013科学发展报告》收录为“中国科学家代表性成果”;被《中国科学院重大成果年报(2012)》收录,入选《中国科学院国际科技合作年度述评》2012年度报告的《国际科技合作重要进展》)
20. Ouyang S#, Gong B#,Li JZ#,Zhao L#,Wu W,Zhang FS,Sun L,Wang SJ,Pan M,Li C,Liang W,Shaw N,Zheng J,Zhao GP,Wang Y*,Liu ZJ*,Liang M*. Structural insights into a human anti-IFN antibody exerting therapeutic potential for systemic lupus erythematosus. J Mol Med (Berl). 2012 July;90(7):837-846. (#共同第一作者)
21. Zhan Z#,Ouyang S#, Liang W,Zhang Z,Liu ZJ*,Huang L*. Structural and functional characterization of the C-terminal catalytic domain of the SSV1 integrase. Acta Crystallogr D Biol Crystallogr. 2012 Jun;68(Pt 6):659-70. (#共同第一作者)
22. Liang W#,Ouyang S#, Shaw N,Joachimiak A,Zhang R,Liu ZJ. Conversion of D-ribulose 5-phosphate to D-xylulose 5-phosphate: new insights from structural and biochemical studies on human RPE. FASEB J. 2011 Feb;25(2):497-504.(#共同第一作者)
23. Qiu XB#*,Ouyang S#, Li CJ,Miao S,Wang L,Goldberg AL*. hRpn13/ADRM1/GP110 is a novel proteasome subunit that binds the deubiquitinating enzyme,UCH37. EMBO J. 2006 Dec 13;25(24):5742-53. (#共同第一作者,已被引用达149次)
24. Yin XQ#,Ouyang S#, Xu WM*,Zhang XP,Fok K.L.,Zhang JP,Miao SY,Chan HC,and Wang LF. YWK-II as a novel Go protein-coupled receptor for Müllerian Inhibiting Substance in cell survival. J Cell Sci. 2007 May 1;120(Pt 9):1521-8. (#共同第一作者)
出版论著
1. 欧阳松应,刘志杰。重要天然免疫系统信号分子STING的结构与功能研究取得重要进展.《2013科学发展报告》,科学出版社,ISBN: 978-7-03-036755-6/N.497
2. 参编《生物信息学方法指南》一书(科学出版社,ISBN 7-03-014465-1,2005年2月)。
(资料来源:欧阳松应研究员,2017-02-23)