2020年1月7日,Cell Reports杂志在线发表了高璞课题组与高光侠课题组合作的研究论文“Molecular Mechanism of RNA Recognition by Zinc-finger Antiviral protein”。该研究工作解析了锌指抗病毒蛋白ZAP N端抗病毒主要功能域与富含CG二核苷酸的单链RNA复合物的高分辨率晶体结构,揭示了锌指抗病毒蛋白ZAP识别单链RNA中CG二核苷酸、单独鸟嘌呤核苷酸以及单独胞嘧啶核苷酸的分子基础。
锌指抗病毒蛋白ZAP最早由高光侠研究员于2002年报道,是对于小鼠白血病病毒的复制具有抑制作用的一种宿主因子。随后的研究发现ZAP特异性抑制多种疾病相关病毒在宿主细胞内的复制,包括艾滋病病毒、埃博拉病毒等。ZAP特异结合病毒的靶RNA序列,干扰靶mRNA的翻译起始。随后ZAP招募细胞内一系列RNA降解机器,包括脱polyA酶PARN、脱帽酶Dcp2、RNA解旋酶p72、核酸外切酶复合体Exosome等降解病毒mRNA。ZAP发挥其抗病毒功能的结构域主要是N端的200多个氨基酸,其晶体结构在2012年时已经被解析,是一种含有四个CCCH类型锌指结构的蛋白分子。然而,ZAP识别RNA的序列特征却一直不清楚。根据2017年的一篇Nature文章报道,ZAP能够特异性识别富含CG的RNA序列。那么ZAP是如何识别CG二核苷酸?除CG二核苷酸之外,ZAP是否还能结合其他的RNA元件呢?再有,ZAP 的主要抗病毒功能域仅有200多个氨基酸,是如何协调多个下游蛋白发挥抗病毒作用的?为回答这些科学问题,研究人员开展了大量的ZAP与RNA复合物结构的研究。
首先,研究人员对不同ZAP蛋白的截短体和不同序列、不同长度富含CG二核苷酸的单链RNA复合物进行了大量的晶体筛选, 最终得到了分辨率为2.19? 的ZAP蛋白N端结构域(NZAP)与6-nt(CGUCGU)单链RNA复合物的晶体结构。在复合物结构中G2-U6五个核苷酸来自一条RNA链结合在一个NZAP分子上,C1胞嘧啶核苷酸结合在NZAP对称分子的一个结合口袋中。与单独蛋白NZAP结构相比,NZAP与RNA复合物结构仍是采取“tractor-like”的构象。通过将复合物结构和单独蛋白结构叠加发现,结合RNA的四个锌指结构的构象都发生了大的变化。NZAP与RNA间的相互作用主要是与RNA上核苷酸碱基间的相互作用,而非磷酸核糖骨架,并且这些碱基都是插入到NZAP一些带正电荷的氨基酸形成的结合口袋中,这个特点也决定了NZAP只能结合单链RNA,而不能与高级结构的RNA形成相互作用。其中C4G5二核苷酸主要结合在ZAP蛋白的第二个锌指区域处,C1主要结合在第三和第四个锌指区域之间,G2主要结合在第三个锌指区域处,并且这三个区域结合的核苷酸都是特异性的。
基于该结构,研究人员通过ZAP蛋白与不同序列单链RNA的ITC结合实验,筛选到优选的ZAP结合RNA motif,即C(n7)G(n)CG,并通过抗病毒实验确证了该motif的重要性。随后,研究人员在ZAP蛋白上构建了一系列突变,并通过EMSA和抗病毒功能分析,确证了ZAP上的这些位点对RNA结合和抗病毒功能的重要性。
同时研究者还发现,如果RNA上存在多个ZAP-binding motif,RNA可同时结合多个ZAP分子。由此可以推测,多个ZAP分子结合在同一条mRNA上,分别招募下游的不同细胞因子,协同发挥RNA降解功能。
该工作是由中科院生物物理研究所高璞课题组和高光侠课题组共同合作完成。高璞研究员和高光侠研究员为本文的通讯作者。高璞课题组的博士研究生骆秀和高光侠课题组的副研究员王新路为文章的共同第一作者。上海光源在晶体衍射数据收集过程中给予了帮助,生物物理研究所平台的陈媛媛博士在ITC实验方面给予了大量的帮助。该工作得到了国家重点研发计划、中国科学院B类战略性先导科技专项、国家自然科学基金的资助。
文章链接:https://www.cell.com/cell-reports/fulltext/S2211-1247(19)31639-0
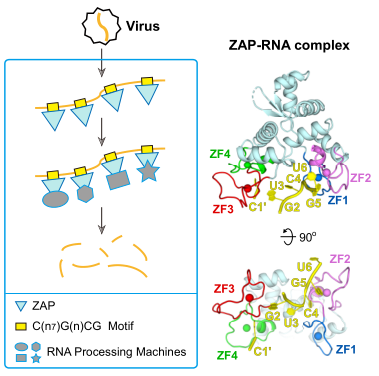
图一:锌指抗病毒蛋白ZAP N端结构域与单链RNA的复合物结构
(供稿:高璞研究组)
 附件下载:
附件下载: