基于单分子定位的超分辨显微成像技术PALM打破了光学衍射极限,于2014年获得了诺贝尔化学奖。相对于目前广泛使用的其它超分辨成像技术而言,该技术具有最高的空间分辨率(~20 nm),因此在生物学中带来了广泛的应用。但是由于该技术需要成千上万张原始图片来重构一张超分辨图像,时间分辨率低,在活细胞中应用该技术面临挑战。
另外,受现有光控荧光蛋白的限制,观察发育过程中超早期结构成像也是目前超高分辨率成像面临的另一挑战。用荧光蛋白标记发育生物具有非入侵,低毒害,低背景等特点,但由于荧光蛋白的折叠和成熟以及累积需要时间,其荧光信号的出现往往滞后于发育中某些早期事件的发生。更早发光的荧光蛋白往往能捕捉发育中更早出现的结构或事件,也使得依据荧光信号进行的定量分析和数据解释变得更加精确和可靠。但目前,在超分辨成像中应用较广的光转化荧光蛋白均不具有发光早的特性。
针对超分辨成像中的这两个问题,徐涛院士研究组和徐平勇课题组合作,开发了一个新型光转化荧光蛋白探针pcStar和一种新型活细胞超分辨成像方法Quick-SIMBA。与合作团队前期发表的被广泛使用的mEos3.2 (Nature Methods,2012) 相比较,pcStar荧光蛋白探针具有发光早,光转化效率高等特点,有利于提高单分子超分辨成像技术的时间分辨率和标记密度,同时可以应用于对短寿命生物分子/结构的超分辨成像。应用新一代单分子定位超分辨成像探针pcStar,课题组成员在细菌,真核细胞系,胎鼠神经干细胞以及果蝇胚胎中均实现了超早期标记。Quick-SIMBA是在徐平勇课题组前期发展的单分子定位超分辨技术SIMBA (Cell Research,2017) 的基础上,结合pcStar探针、sCMOS相机和改进算法提出的新一代活细胞超分辨成像方法。该技术在目前活细胞单分子定位成像技术中具有最高的时空分辨率(0.1-0.25 s, 50 nm),能够很好解析活细胞中的密集管状内质网(图1),这一结构曾因传统成像的时空分辨率不足,而被长时期认为是连续的片状结构。另外,结合pcStar和SIMBA成像技术,课题组成员对果蝇胚胎发育中的超早期结构进行了标记和超分辨成像解析,为该结构的发育形成过程提供了新的证据和视角。

图1:Quick-SIMBA 解析的内质网 (pcStar 标记) 超分辨结构
这一研究成果于10月2日在Nano letters上在线发表,并入选该杂志2020年第四期封面论文,题为 “Fast super-resolution imaging technique and immediate early nanostructure capturing by a photo-convertible fluorescent protein”。该研究工作由中科院生物物理所、中科院计算所、中科院遗传发育所等机构合作完成。生物物理所副研究员张名姝,博士研究生付志飞和副研究员李常青为本文的共同第一作者,徐涛院士和徐平勇研究员为本文的共同通讯作者。徐涛院士领衔的仪器研发团队近年来致力于显微成像仪器设备和技术方法的研究和开发,先后研制出偏振单分子干涉成像、冷冻单分子定位成像以及超分辨光电融合成像系统,开发了多种新的超分辨显微成像方法。徐平勇课题组主要致力于发展多种用于超分辨成像如PALM/STORM、SOFI、SIM等的探针,并基于探针的光化学特性发展新的成像方法如贝叶斯单分子超分辨成像方法SIMBA等,来提高超分辨成像的时空分辨率。该工作是徐涛院士和徐平勇课题组合作产生的又一个成像探针和方法相结合的超分辨成像研究成果。
该研究得到了科技部重点研发计划、国家自然科学基金和中科院先导专项等项目的大力支持。
文章链接:https://pubs.acs.org/doi/10.1021/acs.nanolett.9b02855
2019年10月 5日,《Angewandte Chemie International Edition》期刊以“Hot Article”的形式发表了王江云课题组题为 “S-click reaction for isotropic orientation of oxidases on electrodes to promote electron transfer at low potentials” 的研究文章。文中报道了该课题组开发的基于基因密码子扩展及新型生物正交反应“S-Click”方法改造氨基酸氧化酶,成功实现了氨基酸的快速、实时、精准的电化学检测。
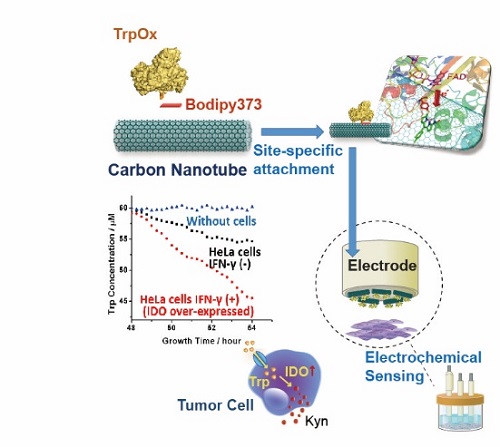
图1. 基于基因密码子扩展的氨基酸生物电化学传感器的示意图
氨基酸是重要的生理生化代谢与细胞信号分子,异常氨基酸代谢导致许多严重疾病,因此实时氨基酸分析对医学、诊断及生命科学具有重要意义。随着生命科学的不断发展,在Cell, Science, Nature等国际顶级期刊上关于氨基酸在细胞代谢,基础病理方面的重要性研究日渐增多,例如色氨酸的代谢,与精神疾病,免疫疾病,以及癌细胞的免疫逃逸都具有重要关联。此外,色氨酸,甘氨酸,精氨酸,丝氨酸的代谢与肿瘤发生发展具有重要关系,已成为重要的精准药物靶点。目前对于氨基酸的检测主要有光谱,液相色谱,酶联显色反应等,上述手段都难以对人体体液中的氨基酸实现实时,动态的分析,也正因为缺乏一种对体液中氨基酸的快捷,实时,灵敏准确分析的手段,不但限制了对不同氨基酸与人类健康关系的进一步研究,而且错失了通过对这种重要生理代谢基础物质的监控来早期预警健康状况的机会。
改进酶电化学生物传感器(EEB)的重点之一是改善酶和电极之间的电子传递。在EEB中使用电子介体是一种改善电子传递的常用方法,但使用电子介体通常会导致相对于酶的原始氧化还原电位增加的过量电位,并且氧化还原介质通常是非选择性的,不仅促进了电极和蛋白质之间的电子转移,也促进了各种干扰分子的电子转移。此外,电子介体在活体分析领域的应用也存在诸多限制。利用纳米材料增强酶电极的电子转移为第3代生物传感器的实现做出了巨大贡献,但酶相对于电极表面的随机取向导致电子转移效率的较大变化。本研究通过基于基因密码子扩展的非天然氨基酸插入技术,位点特异性的将巯基苯丙氨酸(TF)插入到酶特定位点中,TF的巯基通过我们发展的新型生物正交“S-Click”反应与连接分子(Bodipy373)的氯苯基团特异偶联,而连接分子通过π-π stacking组装到碳材料表面,实现不同氨基酸氧化酶在碳材料电极表面的定点偶联(如图2所示)。本团队开创的定点偶联体系(S-Click, 图2C),相比于目前主流的基于叠氮基-炔烃基“点击化学”的偶联体系,具有更好的反应活性与生物相容性,更符合开发可穿戴设备的需求。基于该技术制备的色氨酸氧化酶电极展现出了更高更均匀的酶负载,同时在电化学测试中也展现出了极低催化电位的色氨酸直接生物电催化。(图3)
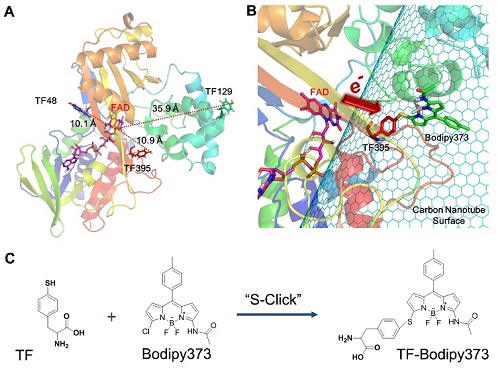
图2:为TrpOx-395TF-Bodipy373形成过程示意图:其中A)TrpOx的结构和TF插入的位置,并显示了TF和FAD之间的距离;B)为TrpOx从TF395位点通过Bodipy373接头到碳纳米管表面的连接示意图;C)为S-Click反应的示意图(S代表TF中的硫原子,Cl代表Bodipy373中的氯原子)。
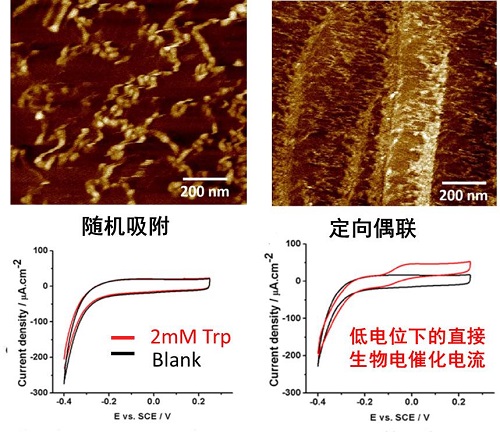
图3:蛋白修饰HOPG电极的原子力显微镜图像以及电化学循环伏安图(CV)
利用基于基因密码子扩展的氨基酸生物电化学传感器,团队进一步在血液样品及癌细胞培养体系中对色氨酸,甘氨酸进行了精准的实时原位动态检测(图4)。
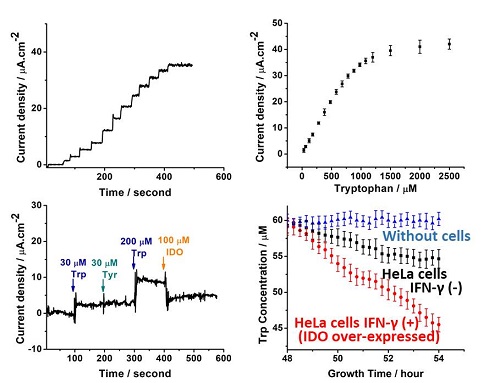
图4:通过S-click定向偶联制备的色氨酸氧化酶电极的时间电流曲线表征,以及在在血液样品及癌细胞培养体系中对色氨酸的实时原位动态检测
该工作由中国科学院生物物理研究所王江云课题组完成。王江云研究员为本文的通讯作者,夏霖副研究员为本文的第一作者。该工作得到了国家自然科学基金、重点研发计划、中科院重点部署项目以及深圳市“三名”工程的资助。
文章链接: https://doi.org/10.1002/anie.201909203
转座子(transposon)由冷泉港实验室Barbara McClintock(诺贝尔奖)首先在玉米中发现【1】。转座子又被称为“跳跃基因”,类似于内源性病毒,能够在宿主基因组中“复制和粘贴”自己的DNA,以达到其自我“繁殖”的目的。转座子的“跳跃”可能会产生基因组不稳定性,并导致动物不孕不育。有多种调控机制沉默转座元件并维持基因组完整性,例如如组蛋白修饰和DNA的甲基化等。为了抵抗转座子,动物的生殖系统进化出了一类小非编码RNA——piRNA(Piwi-Interacting RNA)——来严格调控转座子的表达【2】。概念上,真核生物的piRNA通路在功能上类似于原核生物的CRISPR系统。
piRNA簇(piRNA cluster)表达的piRNA长度大约在24到31nt之间,通过与PIWI家族蛋白(Argonaute家族蛋白的一个亚家族)形成piRISC复合物(piRNA induced silencing complex)而起作用。piRISC复合物能在转录(TGS,Transcriptional gene silencing)和转录后水平的沉默转座子(PTGS,Post-transcriptional gene silencing)【3】。PTGS主要通过“乒乓循环”(ping-pong cycle)在切割piRNA靶标的同时产生更多的新生piRNA,从而形成类似“先天免疫系统”的正反馈,在细胞质层面降解转座子RNA。TGS是在转录水平沉默目标转座子——最终结果是诱导转座子插入位点形成组成型异染色质。转座子的沉默通常跟组蛋白修饰(H3K9me3)有很强的相关性【3】。目前认为,Piwi/piRNA复合物通过转座子新生RNA招募Panoramix(Panx)【4-5】,并最终导致转座子区域异染色质的形成,该过程需要H3K9me3甲基化转移酶SetDB1/Eggless和H3K4me3去甲基化酶LSD1。
2019年9月30日,中科院生物物理所俞洋团队和上海生化细胞所黄旲团队(现上海交大医学院)在Nature Cell Biology杂志上以长文形式在线发表了题为A Pandas complex adapted for piRNA-guided transcriptional silencing and heterochromatin formation的研究,该工作在俞洋/Hannon之前的工作基础上【4】进一步深入阐明了piRNA介导的转座子异染色质形成的分子机制。该研究发现,生殖细胞特异表达的核转运因子(NXF)家族蛋白dNxf2能与dNxt1(P15)以及Panx形成三元复合物,并通过竞争性结合阻止dNxf1(又叫TAP,是介导mRNA出核的经典接头蛋白)与核孔互作,从而导致了转座子新生RNA在核内的滞留。该文章首次证明PANDAS(Panoramix-dNxf2 dependent TAP/p15 Silencing)复合物的存在,并提出了RNA介导异染色质形成的新理论,既阻断新生RNA出核在调控异染色质过程中起核心作用,并为将来研究其它RNA介导的表观遗传调控提供指导意义(图1)。该文章的发表,也给今年四月份在BioRxiv同时上线的关于Drosophila Nxf2的四篇预印本文章之间的良性竞争画上句号【6-9】。
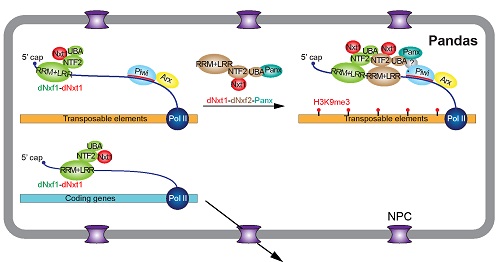
图1. PANDAS复合物调控转座子出核的模型。
本研究中,俞洋课题组充分利用果蝇这一模式生物的优势,综合分析针对piRNA通路的全基因组RNAi筛选结果和flybase中全蛋白组免疫沉淀质谱的大数据,再结合已建立的RNA拴住报告基因筛选体系,首先快速锁定了dNxf2在piRNA/Panx介导的转座子沉默过程中的核心作用。与此同时,俞洋课题组通过和专注于piRNA通路的结构生物学家黄旲课题组的精诚合作,解析了Panx-dNxf2互作结构域的晶体结构。作者通过晶体结构的分析发现,dNxf2的UBA结构域一方面进化出跟Panx直接互作的疏水界面,另一方面也丢失了一般NXF家族蛋白固有的结合核孔复合物的能力(从而解释了为什么dNxf2不能像dNxf1一样介导mRNA出核),为在分子层面进一步理解dNxf2的作用机制奠定了坚实基础(图2)。
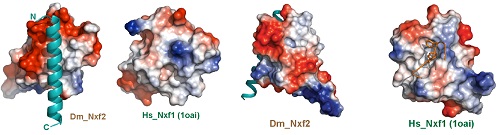
图2. 比较Nxf2型和Nxf1型的UBA结构域。左边,Panx结合面;右边,NPC结合面。
由于dNxf2能跟Panx能形成相互依赖的复合物,dNxf2有着跟Panx类似的诱导基因沉默的功能,即可以通过新生RNA介导基因沉默和异染色质形成。和其它经典piRNA通路蛋白一样,dNxf2的缺失会导致动物体完全不孕不育。有趣的是,小鼠的Nxf2敲除之后在特定条件下也有不育的表型【10】。一系列分子生物学实验和遗传学(co-IP/IP mass spec,GST pull-down,Y2H,GoldCLIP等)也证实dNxf2确实是Panx介导转座子沉默过程中的核心蛋白。但是,作者在研究过程中发现,在dNxf2缺失并一同过表达Panx的条件下,转座子的H3K9me3基本保持不变。具有讽刺意味的是,在此条件下果蝇却是完全不育的,因为转座子仍然是大规模上调的。该结果也直接暗示两点:1. H3K9me3只是异染色质形成的必要非充分条件。2. dNxf2调控转座子沉默的分子机制不仅仅是招募Panx以及下游的甲基化转移酶SetDB1那么简单。
通过大量的文献阅读以及跟研究RNA出核方面的专家(程红)讨论,作者意识到如果仅是dNxf2自身失去核孔复合物结合能力并不足以解释为什么转座子RNA不能出核。dNxf2还需要阻止经典的mRNA出核通路,即通常依赖于mRNA 的5’ Cap结构招募的dNxf1(TAP)复合物。而前人研究也暗示NXF家族蛋白有能力形成异元二聚体,因此,作者大胆假设dNxf2可能通过抑制dNxf1复合物来阻止转座子mRNA的出核。令人兴奋的是,各方面数据包括co-IP,GST pull down,Split luciferase和交联质谱等都充分支持dNxf2:dNxf1能直接互作(图3)。
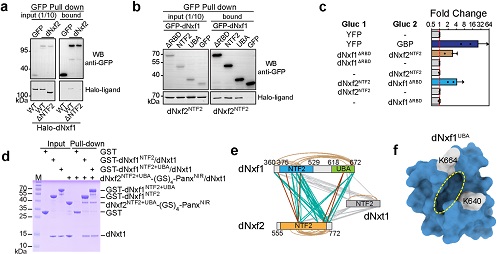
图3. 四方面证据证实dNxf2NTF2跟dNxf1 NTF2+UBA直接互作。
最后,作者通过体外竞争实验和体内瞬时RNA拴住实验为dNxf2抑制dNxf1介导的RNA出核功能这一假说提供了充分的证据(图4)。非常有意思的是,程红课题组最近在哺乳动物的细胞的研究表明Nxf1是RNA聚合酶Pol II有效延伸(transcription elongation)所必须的【11-12】。因此,dNxf2抑制Nxf1的后果很可能不仅仅是阻止转座子RNA的出核,很有能PANDAS会导致Pol II转录延伸的停止。虽然这个理论亟待证实,但是它可以被完美的统一在现有的piRNA介导转座子转录沉默的模型中。
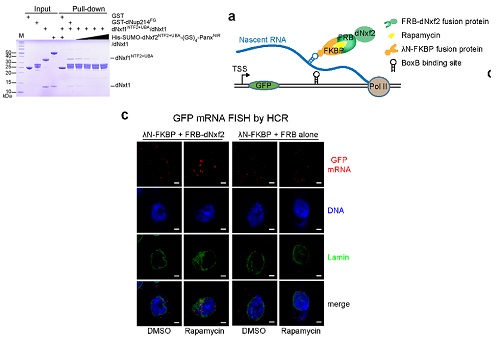
图4. dNxf2通过与Nxf1竞争核孔复合物而阻止新生RNA出核。
本项研究的共同第一作者包括(排名不分先后)中科院生物物理所研究生赵康、苗娜、卢晓华,上海生化细胞所研究生程莎,吉林大学研究生徐平和复旦大学博士后张玉涵。本研究的合作者包括中科院生化细胞所程红研究员、复旦大学麻锦彪教授、北京生命研究所董梦秋研究员和吉林大学万由忠教授。中科院生物物理所俞洋研究员和上海生化细胞所黄旲研究员(现上海交通大学医学院)为共同通讯作者。
文章链接: https://www.nature.com/articles/s41556-019-0396-0
References:
1. McClintock, B. (1953) Induction of Instability at Selected Loci in Maize. Genetics. 38, 579–599
2. Kim, V. N. (2006) Small RNAs just got bigger: Piwi-interacting RNAs (piRNAs) in mammalian testes. Genes Dev. 20, 1993–1997
3. Czech, B., and Hannon, G. J. (2016) One Loop to Rule Them All: The Ping-Pong Cycle and piRNA-Guided Silencing. Trends Biochem. Sci. 0, 324–337
4. Yu, Y., Gu, J., Jin, Y., Luo, Y., Preall, J. B., Ma, J., Czech, B., and Hannon, G. J. (2015) Panoramix enforces piRNA-dependent cotranscriptional silencing. Science. 350, 339–342
5. Sienski, G., Batki, J., Senti, K.-A., D?nertas, D., Tirian, L., Meixner, K., and Brennecke, J. (2015) Silencio/CG9754 connects the Piwi-piRNA complex to the cellular heterochromatin machinery. Genes Dev. 29, 2258–2271
6. The nascent RNA binding complex SFiNX licenses piRNA-guided heterochromatin formation. (2019) bioRxiv 609693; doi: https://doi.org/10.1101/609693
7. Nuclear RNA export factor variant initiates piRNA-guided co-transcriptional silencing. (2019) bioRxiv 605725; doi: https://doi.org/10.1101/605725
8. A Pandas complex adapted for piRNA-guided transposon silencing. (2019) bioRxiv 608273; doi: https://doi.org/10.1101/608273
9. piRNA-guided co-transcriptional silencing coopts nuclear export factors. (2019) bioRxiv 611343; doi: https://doi.org/10.1101/611343
10. Wang, P. J., and Pan, J. (2007) The role of spermatogonially expressed germ cell-specific genes in mammalian meiosis. Chromosome Res. 15, 623–632
11. Chen, S., Wang, R., Zheng, D., Zhang, H., Chang, X., Wang, K., Li, W., Fan, J., Tian, B., and Cheng, H. (2019) The mRNA Export Receptor NXF1 Coordinates Transcriptional Dynamics, Alternative Polyadenylation, and mRNA Export. Mol. Cell. 74, 118–131.e7
12. Co-transcriptional loading of RNA export factors shapes the human transcriptome. (2019) bioRxiv 318709; doi: https://doi.org/10.1101/318709
(供稿:徐平勇研究组)
 附件下载:
附件下载: