Seeing is believing,光学显微镜自1590年由荷兰詹森父子创制伊始,即成为生命科学最重要的研究工具之一。进入21世纪,借助荧光分子,科学家将光学显微镜的分辨率提高了一个数量级,由约一半光波波长(250 nm)拓展至几十纳米,并兴起了超高分辨荧光成像技术,用于“看到”精细的亚细胞结构和生物大分子定位,相关工作荣膺2014年诺贝尔化学奖。
9月9日,Nature Methods杂志在线发表了中国科学院生物物理研究所徐涛院士研究组与科学研究平台纪伟正高级工程师研发团队合作研究论文,题为“Molecular resolution imaging by repetitive optical selective exposure”,为超高分辨光学显微镜家族再添新成员,使显微镜分辨率进一步被突破。该工作提出了一种基于激光干涉条纹定位成像的新技术,并据此研制出新型单分子干涉定位显微镜(Repetitive Optical Selective Exposure, ROSE),将荧光显微镜分辨率提升至3 nm以内的分子尺度,单分子定位精度接近1 nm,可以分辨点距为5 nm的DNA origami(DNA 折纸)结构。
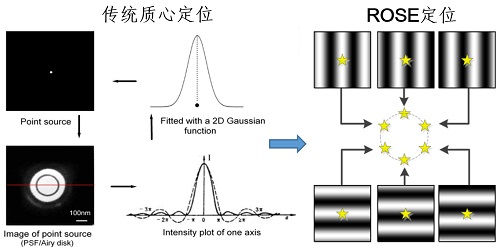
图1 左侧,传统质心拟合定位方法,右侧,ROSE干涉定位方法
所谓干涉定位,是指采用不同方向和相位的激光干涉条纹激发荧光分子,荧光分子的发光强度与其所处条纹的相位有关,该技术即是通过荧光分子强度与干涉条纹的相位关系,来确定荧光分子的精确位置。为降低单分子发光时的闪烁和漂白对亮度和定位精度产生的不良影响,研发团队对显微镜光路进行了创造性地设计,分别为:基于电光调制器的干涉条纹快速切换激发光路,基于谐振振镜扫描的6组共轭成像光路,两种光路的同步实现了高达8 kHz的分时成像,确保在相机的单次曝光时间里把每个单分子发光状态均匀分配给6个干涉条纹,有效避免了荧光分子发光能力波动对定位精度的干扰。
研发团队利用该技术对不同荧光位点间距的DNA origami阵列进行验证测试,证明干涉成像分辨率达到了3 nm的分子水平,可以解析5 nm的DNA origami阵列。后续的功能性实验结果显示,该技术在免疫标记的微管、CCP(clathrin coated pits,网格蛋白有被小窝)以及较致密的细胞骨架成像时展现出良好性能,该技术将为进一步解析精细亚细胞的组分和生物大分子的纳米结构提供有力工具。
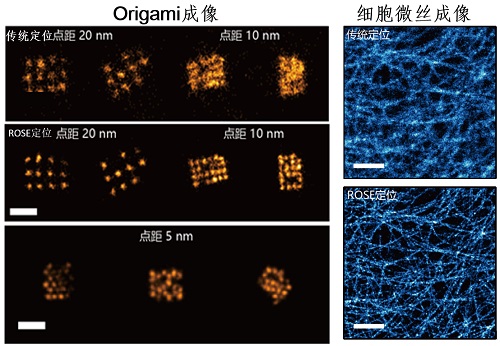
图2左侧,不同荧光位点间距的DNA origami成像,ROSE技术与传统的质心拟合方法进行对比验证。右侧,鬼笔环肽标记的微丝成像,ROSE技术与传统的质心拟合方法进行对比验证。
徐涛院士领衔的仪器研发团队近年来致力于显微成像仪器设备和技术方法的研究和开发,先后研制出偏振单分子干涉成像、冷冻单分子定位成像以及超分辨光电融合成像系统,开发了新的超分辨显微成像算法、探针和技术,申请有多项发明专利,上述成果被广泛应用于细胞生物学相关研究,支撑团队与合作者在该领域取得了系统性成果产出。纪伟正高级工程师所在的生命科学仪器研发中心是根据研究所发展新技术新方法的迫切需求而设立,隶属于科学研究平台,在提供技术服务的同时,聚焦生物显微成像仪器设备的研发与应用推广。
徐涛院士和纪伟正高级工程师为该文章的共同通讯作者,谷陆生、李媛媛、张淑文为共同第一作者。李栋研究员、薛艳红、李尉兴参与了本课题。
该工作受到中国科学院科研仪器设备研制项目、国家重点研发计划、国家自然科学基金以及北京市科技计划等项目的资助。
文章链接: https://www.nature.com/articles/s41592-019-0544-2
(供稿:科学研究平台生命科学仪器研制与服务中心)
 附件下载:
附件下载: