1958 - 1963 中国科学技术大学,生物物理系
1963 - 至今 中国科学院生物物理研究所,研究实习员、助理研究员、副研究员、研究员
1982 - 1984 德国马克斯-普朗克生物化学研究所,生物大分子结构实验室,A.V. Humboldt Research Fellow (洪堡学者)
1988 英国约克大学化学系,蛋白质晶体学实验室,Wellcome Research Fellow
2005 当选中国科学院院士
曾任中科院生物物理研究所副所长,分子生物学研究中心主任,中国生物物理学会副理事长,中国晶体学会副理事长,现兼任《Science China: Life Sciences》主编,《生物化学与生物物理进展》主编。
长期从事蛋白质三维结构及其与功能关系和分子改造研究。早年主要研究蛋白质激素、多肽神经毒素及动植物天然防御蛋白(包括抗真菌、抗病毒、抗肿瘤蛋白),参加我国胰岛素晶体结构研究。主要成果曾获国家自然科学二等奖2次,中科院科技进步一等奖。
研究组近年的研究方向为病原结构生物学,主要研究外源(如病原体感染)和内生(如生理基因突变)病原因素致病,以及宿主应答的蛋白质结构-功能基础,及其基于三维结构的分子机理。疾病的发生与防御及相关免疫反应都有特定的路径,其中重要相关蛋白质及其功能复合物是主要结点,研究在这些致病通路中关键蛋白质及复合物的三维结构及其与致病功能的关系,将为深入解析疾病发生与防御机制提供精确定量的新知识新原理,从而对相关疾病的防治和药物设计具有重要意义,也是全面认识生命活动的一个重要方面。
目前主要研究内容包括下列三个方面:
一、重要病原菌致病关键蛋白质的结构-功能和分子机制。
1. 结核杆菌(MTB)易感性、潜伏性和耐药性相关蛋白质结构-功能基础:发现与鉴定这些MTB致病特性密切相关的蛋白质,解析三维结构,揭示相关作用机理。
2. 革兰氏阴性菌效应蛋白的结构-功能及致病机理:重点研究痢疾杆菌,幽门螺旋杆菌和绿脓杆菌III型,IV型和VI型分泌系统相关的毒力因子和致病相关蛋白的三维结构及其与功能关系。
二、一些内源性疾病的蛋白质结构-功能基础。
1. 内源性基因突变所致神经退行性疾病相关(如AOA1,I型共济失调伴眼动不能症)蛋白质及其病理性作用配体复合物的三维结构与疾病的关系;
2. 内源神经血管瘤和母细胞瘤相关蛋白质CCM系列三维结构及其招募致病通路中重要蛋白质的结构原理研究。近年全面完成人脑海绵状血管瘤(CCM)致病通路中的关键安蛋白CCM 2与其靶蛋白MEKK3激酶调控结构域复合物的三维结构,发现功能单位组装的结构机理,揭示其介导脑血管瘤生成原初过程的分子基础。论文已发表于 Structure.(Structure 2015,23,1087–1096)。
三、宿主细胞应答病原体入侵的免疫反应蛋白质结构-功能基础。
1. 宿主细胞识别病原相关分子模式受体(Pattern Recognition Receptors, PRRs)的结构生物学研究:
Toll-like receptor (TLR) 和NOD-like receptor (NLR) 是识别病原微生物保守分子模式(PAMPs),起始免疫反应的主要功能分子。本研究组重点,研究人类TLR1/TLR2/TLR6识别来自于病原菌细胞膜脂糖的结构基础,揭示这类分子可被不同的TLR异聚二体识别,引发强烈免疫反应的结构机理。同时,结合晶体学与电镜方法,研究新型胞内受体NLR 的结构与功能。
2. 免疫的分子机制研究:
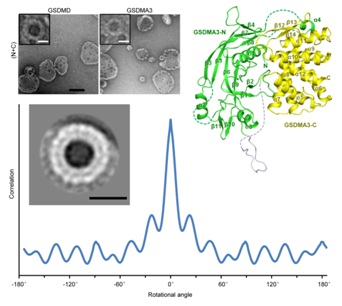
通过合作研究,解析炎性caspase切割的底物蛋白GSDMD三维结构,与功能研究有机结合,揭示天然免疫应答中细胞焦亡的关键分子机制,为多种自身炎症性疾病和内毒素诱导的败血症提供了全新的药物靶点,也为自身炎症性或自身免疫性疾病的治疗提供了全新的思路。论文发表于Nature(2016)。
1. 破解细胞焦亡的关键分子机理: 通过对gasdermin蛋白结构与功能研究的有机结合,首次揭示了gasdermin家族N端结构域具有在膜上打孔进而破坏细胞膜的功能,证明了该家族成员GSDMD是炎性caspase诱导细胞焦亡的直接执行者。研究结果不仅为针对GSDMD开发自身炎症性疾病和败血症的药物奠定了坚实的理论基础,也为后续研究其它gasdermin蛋白在程序性细胞坏死和天然免疫中可能的生理功能开辟了道路。
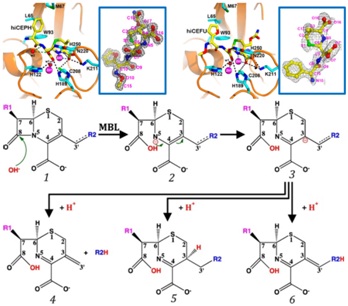
2. 揭示超级细菌金属β-内酰胺酶NDM-1分解头孢菌素类抗生素的分子机理:成功测定NDM-1与头孢类抗生素分解中间产物复合物的高分辨率晶体结构,是国际上首次利用晶体学方法捕捉到头孢类抗生素分解产物中间体的精细三维结构,为NDM-1分解抗生素底物过程中酶-产物中间体复合物的存在提供了坚实的实验证据,也为进一步基于反应限速步骤合理地设计抑制剂提供了充分的理论依据。
1. Ding, J., Wang, K., Liu, W., She, Y., Sun, Q., Shi, J., Sun, H., Wang, D.-C.*, and Shao, F.* (2016). Pore-forming activity and structural autoinhibition of the gasdermin family. Nature 535, 111-116.
2. Wang, X., Hou, Y., Deng, K., Zhang, Y., Wang, D.-C.*, and Ding, J.* (2015). Structural Insights into the Molecular Recognition between Cerebral Cavernous Malformation 2 and Mitogen-Activated Protein Kinase Kinase Kinase 3. Structure 23, 1087-1096.
3. Feng, H., Ding, J., Zhu, D., Liu, X., Xu, X., Zhang, Y., Zang, S., Wang, D.-C.*, and Liu, W.* (2014). Structural and Mechanistic Insights into NDM-1 Catalyzed Hydrolysis of Cephalosporins. Journal of the American Chemical Society 136, 14694-14697.
4. Wang, W., Ding, J., Zhang, Y., Hu, Y.*, and Wang, D.-C.* (2014). Structural insights into the unique single-stranded DNA-binding mode of Helicobacter pylori DprA. Nucleic Acids Research 42, 3478-3491.
5. Li, W.-J., Li, D.-F., Hu, Y.-L., Zhang, X.-E., Bi, L.-J.*, and Wang, D.-C.* (2013). Crystal structure of L,D-transpeptidase LdtMt2 in complex with meropenem reveals the mechanism of carbapenem against Mycobacterium tuberculosis. Cell Research 23, 728-731.
6. Yu, Q., Hu, L., Yao, Q., Zhu, Y., Dong, N.*, Wang, D.-C.*, and Shao, F.* (2013). Structural analyses of Legionella LepB reveal a new GAP fold that catalytically mimics eukaryotic RasGAP. Cell Research 23, 1-13.
7. Xu, X., Wang, X., Zhang, Y., Wang, D.-C.*, and Ding, J.* (2013). Structural Basis for the Unique Heterodimeric Assembly between Cerebral Cavernous Malformation 3 and Germinal Center Kinase III. Structure 21, 1059-1066.
8. Hu, Y., Jiang, F., Guo, Y., Shen, X., Zhang, Y., Zhang, R., Guo, G., Mao, X., Zou, Q.*, and Wang, D.-C.* (2011). Crystal structure of HugZ, a novel heme oxygenase from Helicobacter pylori. J Biol Chem 286, 1537-1544. Selected into “the Encyclopedia of Inorganic and Bioinorganic Chemistry” in 2012 by John Wiley & Sons, Ltd.
9. Gong, Y., Zhu, D., Ding, J., Dou, C.-N., Ren, X., Gu, L., Jiang, T.*, and Wang, D.-C.* (2011). Crystal structures of aprataxin ortholog Hnt3 reveal the mechanism for reversal of 5′-adenylated DNA, Nature Struct Mol Biol.18, 1297–1299.
10. Hu, Y., Gai, Y., Yin, L., Wang, X., Feng, C., Feng, L., Li, D., Jiang, X.N.*, and Wang, D.-C.* (2010). Crystal structures of a Populus tomentosa 4-coumarate:CoA ligase shed light on its enzymatic mechanisms. Plant Cell 22, 3093-3104.
(资料来源:王大成院士,2019-11-22)
王大成 院士 研究员 博士生导师
研究方向:结构生物学-蛋白质三维结构及其与功能关系和分子机理
电子邮件:dcwang@ibp.ac.cn
电 话:010-64888547
通讯地址:北京市朝阳区大屯路15号(100101)
英文版个人网页:http://english.ibp.cas.cn/sourcedb/rck/EN_zgkxyys/202005/t20200519_341430.html