一、什么是染色质?什么是核小体?
染色质是人类遗传物质DNA的载体,是维持人类基因组完整性和稳定性的物质基础。核小体是染色质的基本结构单元,是由多种组蛋白、DNA以及表观遗传标记组成的高度动态的结构单元[1]。从结构上看,核小体拥有一个直径 ~11 nm,厚度 ~5.5 nm 的盘状结构,长度为147 bp的DNA以左手螺旋的方式缠绕在四种核心组蛋白H2A、H2B、H3、H4折叠形成的组蛋白八聚体上。在连接组蛋白H1的作用下,核小体包装并折叠成 ~30 nm的染色质纤维,并进一步形成折叠程度更高的染色质[2]。在转录、复制、修复等过程中,折叠染色质处的DNA逐渐打开,确保DNA与各种分子机器发生相互作用,染色质结构的动态变化是细胞实现其生命进程的基础。
二、病毒如何调节宿主细胞染色质?
病毒与宿主之间的相互抗争表现为病毒的入侵和宿主防御。染色质是宿主细胞抵抗病毒操控宿主的屏障之一,高度凝聚的染色质结构破坏DNA的可及性,从而抵御病毒入侵。病毒通过操控宿主细胞的细胞器,包括染色质的动态变化,来对抗宿主细胞的防御。在正常情况下,宿主细胞通过DNA甲基化、组蛋白修饰、组蛋白变体、染色质重塑,以及非编码RNA等调控因子来改变染色质动态[3]。有趣的是,某些病毒蛋白可以模拟这些调节因子的分子结构,影响宿主的染色质动态,破坏宿主细胞防御。这种称为"分子拟态"的策略基于宿主和病毒蛋白之间结构相似性,被病毒广泛用于染色质动态调节。分子拟态通过干扰异染色质的形成、转录沉默、DNA复制和翻译后修饰等,影响宿主细胞的表观遗传进程。
1. 模拟组蛋白修饰序列
病毒模拟组蛋白序列的传统由来已久。一些病毒利用宿主的组蛋白包装自己的基因组,另外一些病毒则自带组蛋白用于基因组包装。病毒编码的组蛋白不同于宿主组蛋白,但可以形成相似的核小体结构[4]。
除了编码完整的组蛋白,许多病毒选择性地编码包含特定组蛋白序列的蛋白质,该序列可以被宿主细胞识别,并发生翻译后修饰[5]。这种病毒蛋白与组蛋白尾巴存在显著的序列相似性,如位于组蛋白H3的氨基端的ARTK和ARKS序列就存在许多种翻译后修饰。甲型流感病毒H3N26亚型中的非结构蛋白1(NS1)包含一个羧基端ARSK模体,类似于组蛋白H3的ARKS序列。PAF1C(聚合酶相关因子1复合体)和染色质重塑因子Chd1都结合NS1中的ARSK序列,促进病毒的转录或mRNA剪接[6]。SARS-CoV-2蛋白ORF8也含有一个ARKS序列,并可以靶向结合H3K9乙酰转移酶KAT2A。感染严重急性呼吸综合征冠状病毒2(SARS-CoV-2)的细胞会出现基因抑制标记物(H3K9me3和H3K27me3)水平增加和激活标志物(H3K27ac)水平降低,表明ORF8可以通过调节多种组蛋白修饰来降低DNA可及性。进一步的分析揭示了组蛋白模拟在SARS-CoV-2转录反应中的关键作用,因为去除ARKS序列的ORF8使病毒基因组的拷贝数降低[7]。丁型肝炎病毒(HDV)编码的HDV抗原含有在组蛋白 H3氨基端序列KXXR序列。HDV还采用组蛋白模拟策略来促进染色质重塑复合物的招募,完成HDV的复制[8]。
2. 模拟精氨酸指(Arginine finger)
精氨酸指是由几个连续的精氨酸组成的短序列,能够与核小体表面形成的酸性斑块(H2A-H2B酸性残基)选择性结合。非组蛋白通过精氨酸指结合到核小体酸性斑块,是细胞常用的一种染色质锚定机制。病毒蛋白模拟的精氨酸指能够有效结合核小体酸性斑块,将病毒黏附于染色质[9]。在所有已报道的与核小体相互作用的病毒蛋白中,卡波西肉瘤相关疱疹病毒(KSHV)的 LANA、巨细胞病毒(hCMV)的IE1以及骨髓灰质炎病毒(SPV)的Gag 通过精氨酸指状结构与完整的核小体相互作用[10]。
3. 模拟连接组蛋白
腺病毒(Ad)蛋白pVII 是一类富含碱性氨基酸的蛋白质,模拟连接组蛋白H1,与病毒DNA结合形成类似于核小体的"串珠结构"。pVII除了结合病毒DNA,还可以在linker DNA处结合宿主核小体,从而抑制宿主基因组上H2AX的积累和DNA损伤反应。附着于染色质不仅确保可以结合不同的病毒,还可以改变高阶染色质结构动力学[11]。
4. 模拟组蛋白伴侣
精氨酸指与酸性斑块之间的相互作用主要发生在完整的核小体中。最近,中科院生物物理所周政组对EB病毒(EBV)蛋白BKRF4的研究表明,BKRF4可以与部分未折叠的核小体结合。有趣的是,BKRF4通过"triple-anchor"结合模式与H2A-H2B二聚体相互作用,BKRF4-H2A-H2B的相互作用会诱发核小体DNA的解缠绕以及组蛋白八聚体结合位点的暴露。因此,BKRF4倾向于在DNA断裂处与部分未折叠的核小体相互作用,并干扰RNF168的招募,从而阻止DDR信号传播。BKRF4与H2A-H2B或H3-H4的结合方式表现出其组蛋白伴侣功能,表明BKRF4采用组蛋白伴侣的模拟机制来干扰核小体的组装以及下游蛋白的招募[12]。
5. 模拟DNA
白斑综合征病毒蛋白ICP11富含酸性氨基酸的蛋白质,模拟DNA与碱性的组蛋白发生作用。ICP11与组蛋白H2A、H2B、H3 和H2A.X相互作用。ICP11可能干扰核小体组装,阻止H2A.X完成其修复DNA双链断裂的关键功能。能够模拟DNA的分子可以起到组蛋白伴侣的作用,通过占据组蛋白上的DNA结合位点调控其DNA结合活性,从而影响染色质的动态[13]。
6. 模拟转录因子
研究表明,染色质相分离的过程也会影响染色质动力学,转录因子是介导染色质相分离的重要因子。最近一项研究表明,EB病毒蛋白EBNA2作为病毒蛋白的转录因子,可以通过相分离重组染色质拓扑结构,并招募组蛋白乙酰转移酶p300在特定区域进行组蛋白H3K27乙酰化[14]。EBNA2与转录因子之间的序列相似性表明,病毒模拟转录因子介导相分离,进而实现染色质重组。
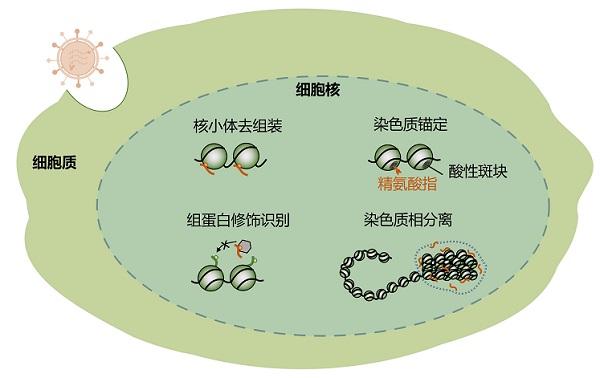
病毒通过分子拟态调控染色质动态的主要模式
病毒蛋白通过模拟组蛋白伴侣调控核小体去组装,模拟精氨酸指实现染色质锚定,模拟组蛋白序列调节细胞对组蛋白修饰的识别,模拟转录因子调节染色质相分离。橙色碎片代表病毒蛋白。
三、 总结与展望
人类与病毒之间的博弈是一场"持久战",人类只有知己知彼,才能在博弈中抢占先机。而不同的病毒在侵染宿主的过程中采用了多种策略,它们调控宿主染色质结构和功能的方面还有哪些不为人知的方法?仍然留下了很多未解之谜,值得我们在未来的研究中潜心探索!
参考文献
1. Luger K, Maer A W, Richmond R K, et al. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature, 1997, 389(6648): 251-260
2. Song F, Chen P, Sun D, et al. Cryo-EM study of the chromatin fiber reveals a double helix twisted by tetranucleosomal units. Science, 2014, 344(6182): 376-380
3. Zhou K, Gaullier G, Luger K. Nucleosome structure and dynamics are coming of age. Nat Struct Mol Biol, 2019, 26(1): 3-13
4. Liu Y, Bisio H, Toner C M, et al. Virus-encoded histone doublets are essential and form nucleosome-like structures. Cell, 2021, 184(16): 4237-4250.e19
5. Strahl B D, Allis C D. The language of covalent histone modifications. Nature, 2000, 403(6765): 41-45
6. Schaefer U, Ho J S Y, Prinjha R K, et al. The "histone mimicry" by pathogens. Cold Spring Harb Symp Quant Biol, 2013, 78: 81-90
7. Kee J, Thudium S, Renner D M, et al, SARS-CoV-2 disrupts host epigenetic regulation via histone mimicry. Nature, 2022, 610(7931): 381-388
8. Abeywickrama-Samarakoon N, Cortay J C, Sureau C, et al. Hepatitis Delta Virus histone mimicry drives the recruitment of chromatin remodelers for viral RNA replication. Nat Commun, 2020, 11(1): 419
9. Barbera A J, Chodaparambil J V, Kelley-Clarke B, et al. The nucleosomal surface as a docking station for Kaposi's sarcoma herpesvirus LANA. Science, 2006, 311(5762): 856-861
10. Lesbats P, Serrao E, Maskell D P, et al. Structural basis for spumavirus GAG tethering to chromatin. Proc Natl Acad Sci USA, 2017, 114(21): 5509-5514
11. Avgousti D C, Herrmann C, Kulej K, et al. A core viral protein binds host nucleosomes to sequester immune danger signals. Nature, 2016, 535(7610): 173-177
12. Chen J, Lu Z, Gong W, et al. Epstein-Barr virus protein BKRF4 restricts nucleosome assembly to suppress host antiviral responses. Proc Natl Acad Sci USA, 2022, 119(37): e2203782119
13. Wang H C, Wang H C, Ko T P, et al. White spot syndrome virus protein ICP11: a histone-binding DNA mimic that disrupts nucleosome assembly. Proc Natl Acad Sci USA, 2008, 105(52): 20758-20763
14. Yang Y, Ye X, Dai R, et al. Phase separation of Epstein-Barr virus EBNA2 protein reorganizes chromatin topology for epigenetic regulation. Commun Biol, 2021, 4(1): 967
作者简介
王亚静

中国科学院生物物理研究所,中国科学院大学硕士研究生
肖淑敏

中国科学院生物物理研究所,中国科学院大学硕士研究生
周政
中国科学院生物物理研究所研究员,中国科学院大学岗位教授,主要从事染色质动态的功能及机制研究。
科学寄语
知己知彼,百战不殆!
 附件下载:
附件下载: