柳振峰课题组发表光合作用状态转换机制研究方面的最新进展
2015年4月17日,Plant Cell 期刊在线发表了中国科学院生物物理研究所柳振峰课题组报道的植物光合作用状态转换磷酸酶(TAP38/PPH1)底物识别机制的研究成果,题为“Structural Mechanism Underlying the Specific Recognition between the Arabidopsis State-Transition Phosphatase TAP38/PPH1 and Phosphorylated Light-Harvesting Complex Protein Lhcb1”。
状态转换(state transition)是藻类和陆生植物在弱光条件下响应外界光照质量(light quality)变化的一种巧妙的适应调控机制,对于植物在多变的光照条件下优化光合作用效率具有重要的作用。植物光合作用中需要两个光系统(光系统I和II)的平衡运转来实现光驱动的能量物质生成过程。在状态转换可逆的调控过程中,植物利用一对分别被称为Stt7(或STN7)和TAP38(或PPH1)的叶绿体蛋白激酶和磷酸酶来调控天线复合物LHCII的磷酸化水平,实现光能在两个光系统间的动态平衡分配。关于LHCII磷酸酶的研究始于上世纪80年代,而其具体的结构特征和作用机制三十多年来一直是个谜。2010年,植物分子遗传学家(德国Leister组和瑞士Goldschmidt-Clermont组)分别独立发现TAP38/PPH1对于LHCII的去磷酸化和状态转换的逆过程是必需的,然而其是否能直接作用于磷酸化的LHCII及其分子间的相互识别方式都还有待进一步研究阐明。
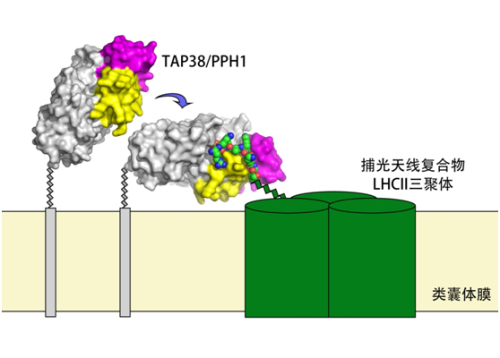
柳振峰课题组通过生物化学分析方法证明了状态转换磷酸酶TAP38/PPH1可特异性地直接作用于磷酸化LHCII并能够高效地将其去磷酸化,而对于光系统II核心亚基磷酸化蛋白的催化活性则较弱。经过多年的努力,课题组解析了TAP38/PPH1基质侧的核心结构域蛋白本身,及其与磷酸化天线蛋白Lhcb1的氨基端多肽复合物的高分辨率晶体结构(图1)。通过对两个结构的分析和比较的结果揭示了TAP38/PPH1得以特异性地识别磷酸化LHCII的分子机制。研究发现了TAP38/PPH1中存在两段特有的氨基酸序列和相应的结构模(Motifs A和B,图1黄色和紫色部分),它们参与形成两个特异的底物结合和识别位点,在底物识别过程中发挥着关键的作用。此外,TAP38/PPH1与pLhcb1多肽复合物的晶体结构也是2C型蛋白磷酸酶家族中首次被报道的该类型磷酸酶与磷酸化多肽底物复合物的结构,为深入理解2C型蛋白磷酸酶的催化过程反应机制提供了高精度的分子间相互作用细节。
图1、状态转换磷酸酶TAP38/PPH1与捕光天线复合物LHCII相互作用的示意图。色彩模式:灰色,TAP38/PPH1的催化结构域及预测的一段跨膜螺旋;黄色,Motif A;紫色,Motif B;绿色柱体,捕光天线复合物LHCII的膜内部分;绿-蓝-红三色球体模型,捕光蛋白Lhcb1的N-末端磷酸化多肽;蓝箭头显示TAP38/PPH1从无底物结合状态到与底物pLhcb1结合的转换过程。
本项研究得到了《光合作用与“人工叶片”》973计划重大科学问题导向项目以及中国科学院战略性先导科技专项(B类)的资助。课题负责人和论文通讯作者为柳振峰研究员究。论文第一作者魏雪鹏(博士研究生)负责实施论文中涉及分子生物学、生物化学、衍射数据收集、结构解析和数据分析总结等一系列的工作。该项工作的顺利开展得到了常文瑞课题组的李梅博士和郭江涛博士的参与和支持,晶体衍射数据收集于上海光源BL17U线站和光子工厂NW12A线站。
全文链接:http://www.plantcell.org/content/early/2015/04/17/tpc.15.00102.full.pdf+html
(供稿:柳振锋课题组)
附件下载: