Genes & Development杂志发表生物物理所许瑞明组
2013年1月1日,美国Genes & Development杂志发表了生物大分子国家重点实验室许瑞明研究组与美国Stony Brook大学Rolf Sternglanz课题组合作的研究成果。该文章题为“Structural Basis for Allosteric Stimulation of Sir2 Activity by Sir4 Binding”。详见网址:
http://genesdev.cshlp.org/content/27/1/64.full.pdf+html 。
Sirtuin家族的蛋白质是一类新颖的去乙酰化酶,它们的催化活性依赖于NAD,这一家族的酶在酵母的转录沉默、高等动物的长寿和衰老以及糖尿病的发生中都有重要的作用。其中酵母Sir2(Silent information regulator 2)蛋白是该家族第一个被发现的成员。Sir2单体时就具有依赖NAD的去乙酰化酶活性,而与Sir4的结合提高了酶活性。2001年许瑞明课题组首次报道了古菌中Sir2同源蛋白的晶体结构(Cell 2001, Cover Story),揭示了这一大类酶的活性中心的组分和结构,找出了底物乙酰化赖氨酸的结合通道,提出了这一类酶的催化机理。
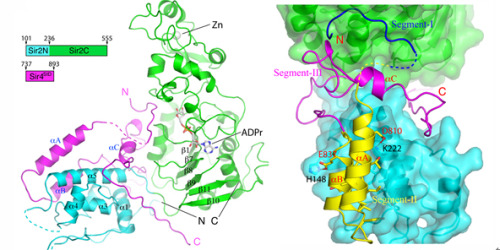
此项最新研究中,我们解析了Sir2和Sir4SID(Sir2 Interaction domain of Sir4)复合体的晶体结构。发现Sir4主要结合在Sir2的N端,通过一个长loop与Sir2 N端结构域及C端催化域的交界处相互作用。进一步研究发现,Sir4通过与Sir2N-Sir2C两个结构域交界处的相互作用,调节Sir2结构域间的构象变化,进而调节Sir2的去乙酰化酶活性。这一结果对我们理解Sirtuin家族去乙酰化酶活性的调节机制具有普遍意义。
该项研究工作得到科技部、国家自然科学基金委员会和中国科学院的资助。
图示 Sir2-Sir4SID相互作用结构示意图(左)及相互作用的主要部位(右)
(供稿:许瑞明课题组)
附件下载: