
生物物理所研究抗病毒蛋白ZAP活性区域
3月11日,国际著名期刊Nature Structural & Molecular Biology发表了中国科学院生物物理所刘迎芳研究组和高光侠研究组合作完成的研究成果——抗病毒蛋白ZAP活性区域的晶体结构与功能研究(Structure of N-terminal domain of ZAP indicates how a zinc-finger protein recognizes complex RNA)。
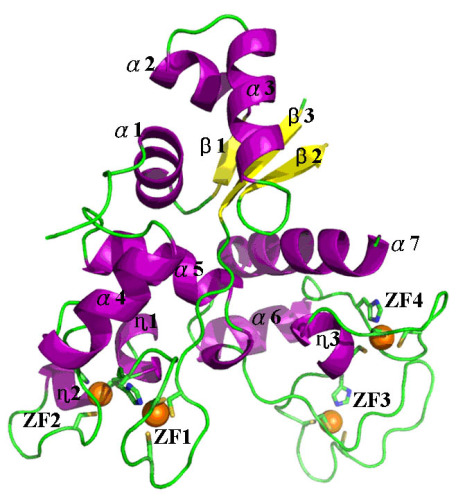
ZAP(Zinc finger Antiviral Protein)是2002年发现的具有抗病毒活性的宿主细胞限制性因子,从细胞中分离得到的ZAP蛋白在体内可以显著抑制特定病毒在宿主细胞中的复制,包括对人类健康造成重大威胁的艾滋病病毒(HIV-1),埃博拉病毒(Ebola virus)等。但是不抑制流感病毒等。同时,ZAP蛋白在天然免疫干扰素途径激活过程中扮演着重要角色。研究表明,ZAP蛋白通过其N端结构域是抗病毒活性的主要区域,该区域能结合病毒mRNA并招募核酸外切酶复合物,行使从3’到5’降解病毒mRNA的功能。另外,在对ZAP蛋白在天然免疫信号通路中所起作用的研究也表明,ZAP蛋白N端结构域对于其结合并激活RIG-I蛋白具有决定作用。目前发现的ZAP能够识别并抑制的病毒mRNA都很大,长度超过500个核苷酸残基;该核酸缩短可能导致不再被ZAP抑制。长期以来,研究人员在抗病毒蛋白ZAP的功能研究过程中一直存有一些疑问:仅仅由254个氨基酸组成N端结构域如何在抗病毒过程中发挥作用的?ZAP蛋白N端结构域的锌指结构具有怎么样的结构特点,从而可以特异性识别较大分子量的病毒RNA?病毒RNA可能具有什么特性可以被ZAP特异性识别并抑制?基于以上问题,我们开展了ZAP蛋白N端结构域的结构生物学研究。
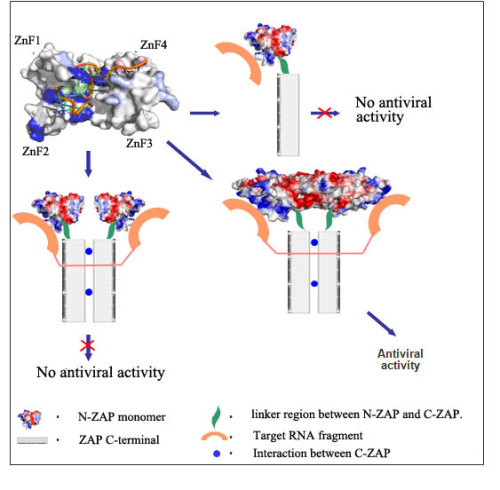
通过对ZAP蛋白活性区域的晶体结构测定工作,我们首次揭示了一种同时含有四个CCCH类型锌指结构的蛋白质分子三维结构,为ZAP蛋白特异识别病毒RNA分子并发挥其抗病毒活性提供了准确的结构基础;通过一系列体内抗病毒实验以及体外与病毒RNA的结合实验对ZAP蛋白N端结构域重要的RNA结合位点进行了验证,发现了ZAP蛋白表面含有一个由四个锌指结构构成的面积约300平方埃的、三维的特异性结合病毒RNA的区域;通过实验发现并证实了ZAP蛋白分子氨基端存在分子间相互作用,这种相互作用会促使ZAP蛋白在体内抗病毒过程中以二体形式识别并结合病毒RNA,这对抗病毒活性中起决定作用。同时,我们发现ZAP能够识别并抑制的RNA不仅可能存在特殊的高级结构,而且还含有两个ZAP结合区域,这就解释了为什么ZAP靶RNA都非常大。
该项工作是由中国科学院生物物理研究所刘迎芳研究员与高光侠研究员所率领的课题组合作完成的。在此过程中,研究人员克服了ZAP蛋白容易发生沉淀及不结晶等障碍,进而进一步完成了一系列后续工作。该项工作主要完成人有刘迎芳课题组的陈守登博士和高光侠课题组的徐义辉博士。其他主要参与人员有:张阔,孙建(刘迎芳课题组);王新路博士(高光侠课题组)等。除此之外,其他一些人员也提供了重要帮助。日本KEK和上海SSRF提供了同步辐射光源支持。该项研究课题得到了科技部,国家自然科学基金委和中国科学院的资助。
 |
 |
|
图1. ZAP氨基端(N-ZAP)三维结构图。 |
图2. ZAP功能模式图 |
(供稿:刘迎芳课题组、高光侠课题组)
附件下载: