生物成像中心携手海光信息:打造冷冻电镜数据处理国产化新里程碑
针对该领域,中国科学院生物物理研究所蛋白质科学研究平台生物成像中心(以下简称“生物成像中心”)与海光信息开展深度合作,以海光K100_AI加速卡为基础,针对冷冻电镜数据处理流程中的关键算法与工具进行国产算力适配迁移。近日,双方联合在海光平台上完成了全面适配与性能优化,包括MotionCor3、IMOD、CTFFIND4和RELION 5等学科主流软件和自研模型DeepETPicker。
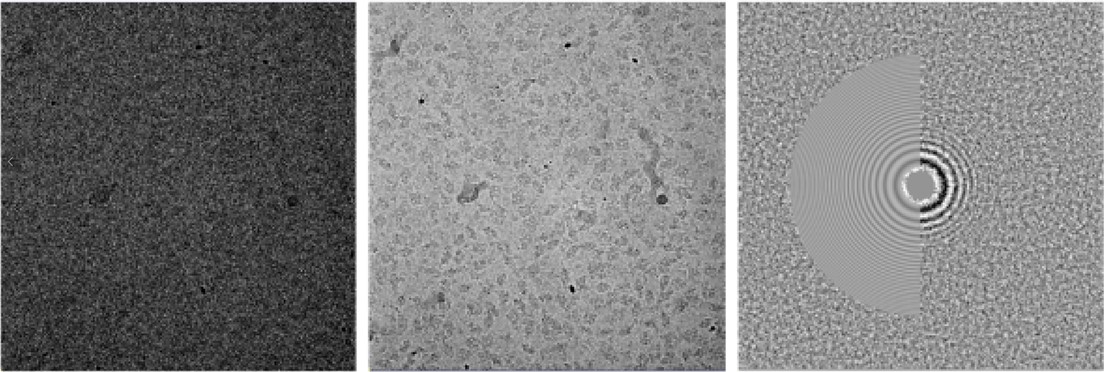
MotionCor3在海光K100_AI进行图像抖动矫正的结果
在冷冻电镜图像处理的首个关键环节——抖动校正(motion correction)中,得益于海光DTK开发套件 优良的生态兼容性,开源的MotionCor3代码实现了直接在海光DCU加速器 上编译与运行。以约1.8 Å分辨率测定的大肠杆菌β-半乳糖苷酶结构图像为样本,系统成功完成了帧间位移校正,并结合CTFFIND4 实现了对比传递函数(CTF)估计与像差修正,确保了多帧平均过程中的剂量加权、傅里叶环匹配等关键计算结果的准确无误——标志着冷冻电镜数据预处理链条在国产算力平台上实现了可靠落地。
在原位冷冻电子断层数据颗粒识别阶段,已适配由生物成像中心与中国科学院自动化研究所联合开发的 DeepETPicker模型。该模型采用弱监督学习方法,可在校正后的显微图像中高效筛选出数百万个蛋白质颗粒。其深度学习模型在迁移至海光K100_AI加速卡 后,通过pycm、scipy源码重构与计算图优化等手段,实现了在 FP32精度下与NVIDIA RTX 4090Ti相当的训练与推理性能,展示了国产AI算力在科研应用场景下的强大潜能。
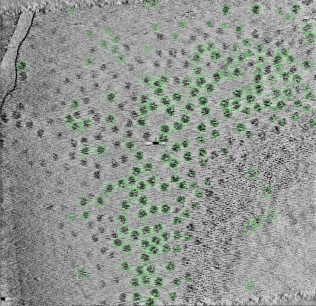
DeepETPicker基于海光K100_AI加速卡推理结果
对于综合性冷冻电镜单颗粒分析软件,海光团队完成了最新版RELION 5的适配与优化。针对三维重构阶段中FFT计算显存规划失败的难题,团队通过“零填充(zero-padding)优化”策略,有效改变平移格点分布、提高FFT规划的稳定性与效率,使三维密度图的后处理结果更加清晰细腻,大大提升了最终模型的分辨率与可解释性。
从精准抖动校正、像差参数估计,到智能颗粒挑选,海光算力可实现自主管线设计的高精度三维重构,更兼顾成熟的全流程套件。冷冻电镜数据处理链如今已在海光平台上实现了自主可控、稳定高效的全流程运行,同时兼顾科研工作的自定义创新需求。这不仅意味着“从使用先进工具”迈向“掌握先进算力”的关键跨越,也标志着我国在生命科学计算领域实现了从算法到算力的真正融合。
未来,中国科学院生物物理研究所生物成像中心将依托海光信息的国产设备支持,继续深入推进冷冻电镜软件生态的国产化与智能化,加速破解生命奥秘,构筑面向生命科学与新药研发的“中国算力”底座,让中国的科研力量在全球结构生物学的舞台上闪耀更亮的光芒。